Els fitxers SDF contenen informació estructural d’una o més molècules químiques.
On this page
Introducció
Els Structured Data Format, que solen tenir la extensió .sdf, són els fitxers que els programes utilitzen habitualment per processar informació estructural química. També poden incloure informació addicional, com ara espectres moleculars.
En principi, qualsevol fitxer de dades estructurals té dos components principals:
- La taula de connexions simplificada (simplified connection table)
- La informació addicional
La notació SMILES descriu la connectivitat atòmica en text, mentre que els fitxers SDF contenen informació estructural més detallada. En SMILES els hidrogens solen ser implícits (per exemple l’aigua es representa com a O en Smiles); en SDF poden ser implícits o explícits (habitualment són explícits).
Tots els fitxers de dades estructurals tenen una taula d’àtoms i una taula d’enllaços.
La taula d’àtoms inclou informació com les definicions isotòpiques i, si està disponible, les coordenades 2D o 3D de l’entorn de cada àtom. Si aquestes coordenades falten, els programes poden calcular-les mitjançant una minimització d’energia per obtenir l’estructura 3D d’un àtom aïllat.
Tipus de fitxers: SDFs i Molfiles
- Molfiles: fitxers de text que contenen informació estructural d’una sola mol·lecula. Solen tenir la extensió
.mol. - SDFs (Structure Data Files): són fitxers que combinen diversos molfiles amb informació addicional sobre els compostos. S’utilitzen molt sovint per compartir biblioteques de dades de compostos.
Què tenen en comú ?
- Tant els Molfiles com els SDFs (Structure Data Files) són fitxers de text que descriuen l’estructura química de molècules:
- àtoms, enllaços, coordenades, càrregues, etc.
- Tots dos segueixen el format desenvolupat per MDL (Molecular Design Limited), de manera que la seva estructura interna és gairebé la mateixa.
- Són formats llegibles per humans i per ordinador, i compatibles amb molts programes de química computacional (com RDKit, PyMol, Avogadro, etc.).
En resum, els Molfiles descriuen una sola molècula; els SDFs amplien aquest format per incloure moltes molècules i dades associades, mantenint la mateixa base estructural.
Estructura dels Molfile/SDF
L’NCI Chemical Resolver és un servei en línia del National Cancer Institutre que permet buscar i obtenir informació química de compostos des de diverses bases de dades obertes (per exemple Pubchem) a partir del seu nom, fórmula o identificador.
El següent text és un fitxer molfile de l’etanol obtingut del NCI Chemical Resolver.
C2H6OAPtclcactv10282509333D 0 0.00000 0.00000
9 8 0 0 0 0 0 0 0 0999 V2000 1.2854 -0.2499 -0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -0.0072 0.5687 -0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -1.1304 -0.3147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 1.3175 -0.8784 0.8900 H 0 0 0 0 0 0 0 0 0 0 0 0 1.3175 -0.8784 -0.8900 H 0 0 0 0 0 0 0 0 0 0 0 0 2.1422 0.4239 -0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 -0.0392 1.1972 0.8900 H 0 0 0 0 0 0 0 0 0 0 0 0 -0.0392 1.1972 -0.8900 H 0 0 0 0 0 0 0 0 0 0 0 0 -1.9857 0.1365 -0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 1 2 1 0 0 0 0 2 3 1 0 0 0 0 1 4 1 0 0 0 0 1 5 1 0 0 0 0 1 6 1 0 0 0 0 2 7 1 0 0 0 0 2 8 1 0 0 0 0 3 9 1 0 0 0 0M END$$$$Tots els molfiles tenen un encapçalament i una taula de connexions (CTAB) amb dos blocs principals: Atom Block i Bond Block.
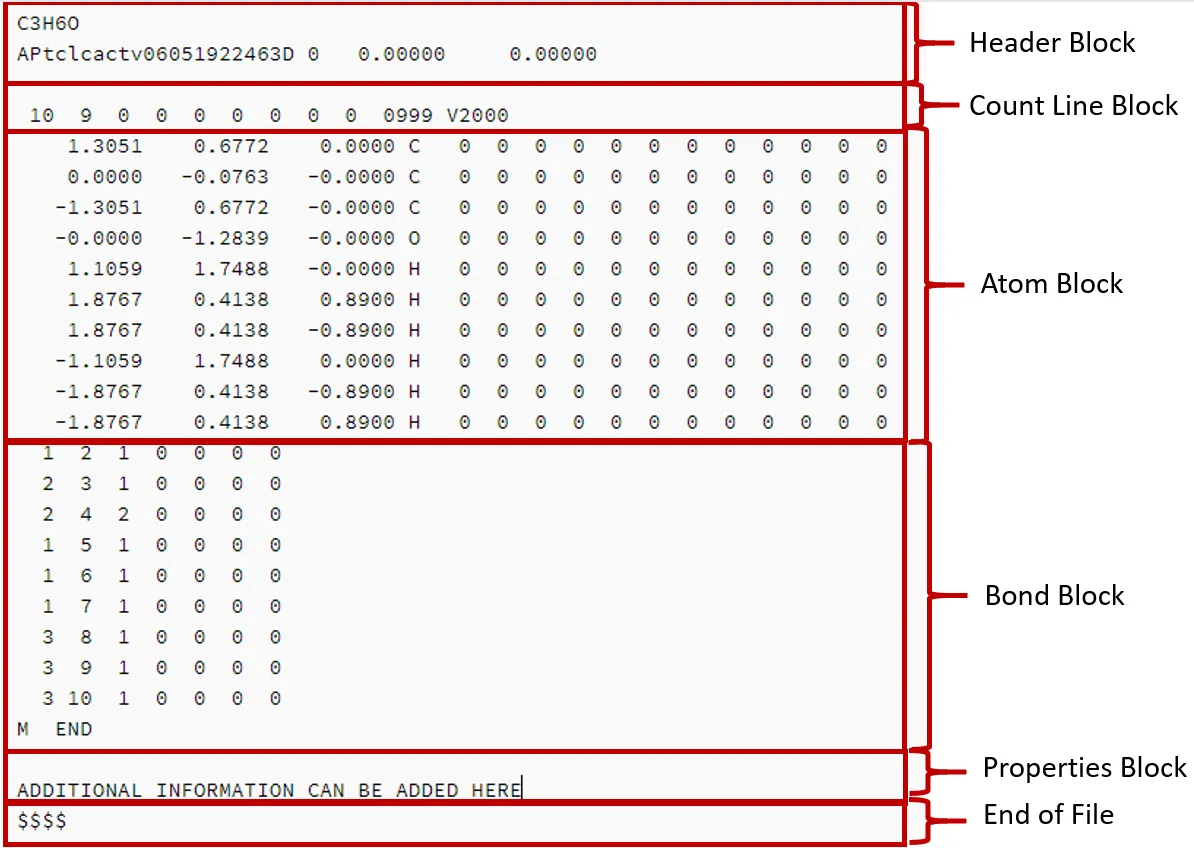
- L’encapçalament té dues o tres línies: la primera indica el nom o fórmula i la segona el programa que el va generar, la data i hora, i si hi ha coordenades 2D o 3D. Opcionalment, pot tenir una linia 3 amb comentaris.
- La línia de recompte (
Count line block) mostra que l’acetona té 9 àtoms i 8 enllaços, i inclou la versió del molfile, en aquest cas V2000. - Després del
Bond Block, hi ha una descripció addicional i camps opcionals que poden incloure propietats físiques, dades experimentals o anotacions de la molècula.
A l’exemple de sota, la fórmula que identifica la mol·lecula és C2H6O, ha estat generat per APtclcactv10282509333D, i el camp de comentaris està en blanc.
Veiem un altre exemple de molfile, de l’acetona, en format imatge per tenir l’esquema complet més clar:
Cercar fitxers sdf a Internet.
Pots obtenir altres compostos des de la web pública de l’NCI Chemical Resolver, amb aquestes opcions de menú:
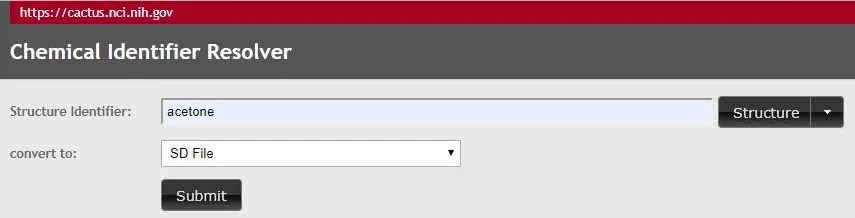
Encara que posi sdf, com que sabem que l’acetona només consta d’una mol·lecula ens serveix com a molfile, coincideix pràcticament tot el contingut.
Taller: Descarregar fitxers sdf amb Python.
Pots obtenir l’acetona a partir d’una URL, la qual cosa és encara més interessant.
Fixa’t que la notació general és
https://cactus.nci.nih.gov/chemical/structure/{NOTACIO_SMILES}/file?format=sdf
On pots reemplaçar {NOTACIO_SMILES} per la notació SMILES de la mol·lecula que vols.
Prepara l’entorn en Python:
Crea un projecte amb uv
uv init demo-sdfsImporta la biblioteca requests:
uv add requestsAquesta llibreria et permet consultar fitxers de text o respostes textuals que provinguin d’un servidor web si la url és vàlida.
Això ho fem amb la funció:
url = "https://cactus.nci.nih.gov/chemical/structure/CCO/file?format=sdf"r = requests.get(url)Posteriorment, els descarregarem agafant la informació consultada (està a la variable r en el nostre cas) i guardant-la en un nou fitxer.
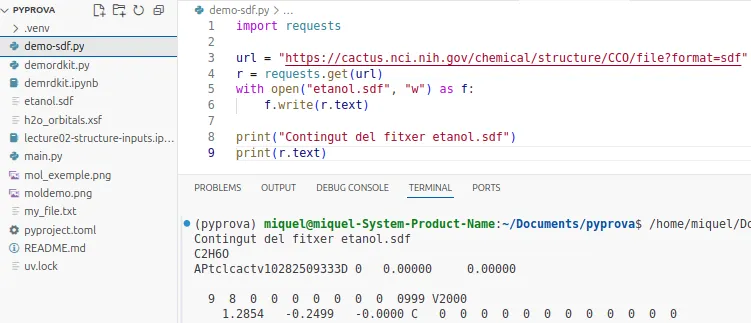
# Agafem les dades extretes d'Internet i les guardem al fitxer.with open("etanol.sdf", "w") as f: f.write(r.text)Ara, modifica el fitxer main.py amb totes les instruccions:
import requests
url = "https://cactus.nci.nih.gov/chemical/structure/CCO/file?format=sdf"# Descarreguem el fitxerr = requests.get(url)# Agafem les dades extretes d'Internet i les guardem al fitxer.with open("etanol.sdf", "w") as f: f.write(r.text)
print("Contingut del fitxer etanol.sdf")print(r.text)Pots comprovar que s’ha creat un fitxer amb el nom etanol.sdf que conté la mateixa mol·lecula que hem analitzat fa una estona.
Aquesta tècnica és ideal per fitxers petits com els molfile i els sdf.
Llegir únicament primeres linies.
Aquí tens un codi per a mostrar únicament les primeres linies del fitxer, per si només t’interessa l’encapçalament i la linia de recompte.
primeres_linies = 4with open("etanol.sdf") as f: for i in range(primeres_linies): linia = f.readline() if not linia: break print(linia.strip())Si vols saber com llegir i escriure qualsevol fitxer de text amb Python consulta Python - File.
Per a fitxers més grans i gestionar control d’errors recomanem Python - urllib3.
Taller: Llegir fitxers sdf amb RDKIT
Si encara no ho has fet, importa la biblioteca rdkit al teu projecte:
uv add rdkitPer automatitzar la tasca, executa aquest programa, que es baixarà alguns fitxers sdf únicament en cas que no els hagis baixat abans (♻️ mesura sostenible ♻️) amb la llibreria nativa os
import osimport requests
# Llista de compostos simplesmolecules = ["water", "caffeine", "aspirin"]
# 📁 Carpeta on guardarem els arxiusos.makedirs("sdf_files", exist_ok=True)
for name in molecules: file = f"sdf_files/{name}.sdf" if not os.path.exists(file): print("Descarregant", name) url = f"https://cactus.nci.nih.gov/chemical/structure/{name}/sdf" data = requests.get(url).text open(file, "w").write(data) else: print(name, "ja existeix")
print("\n✨ Procés complet! Els arxius estan dins la carpeta 'sdf_files'.")Un cop descarregat un fitxer, pots obrir la mol·lecula amb RDKIt, per a mostrar les seves propietats, visualitzar-la… com vas fer a Bio - SMILES
from rdkit import Chemfrom rdkit.Chem import Drawfrom rdkit.Chem import rdMolDescriptors
# Guardem totes les mol·lècules en una llista (sdf pot tenir més d'una)molecules = []# Obrim el fitxer d'exemple amb "with" per assegurar que es tanqui béwith Chem.SDMolSupplier("sdf_files/caffeine.sdf") as suppl: for mol in suppl: molecules.append(mol)
print(f"Número de mol·lecules: {len(molecules)}")
form_mol = rdMolDescriptors.CalcMolFormula(molecules[0])print(f"Fórmula: {form_mol}")Activitat
- Descarrega el SDF de la hormona de la felicitat 😊, la serotonina, des del NCI Chemical Resolver.
- Guarda el fitxer dins de sdf_files/.
- Mostra per pantalla les primeres linies del fitxer i respòn quants àtoms i enllaços té en total la hormona.
- Llegeix la molècula amb RDKit.
- Mostra la fórmula molecular i la notació SMILES.
- Guarda una imatge 2D de la molècula.
import osimport requestsfrom rdkit import Chemfrom rdkit.Chem import Drawfrom rdkit.Chem import rdMolDescriptors
# 1 Descarrega el SDF de la serotoninaurl = "https://cactus.nci.nih.gov/chemical/structure/serotonin/sdf"response = requests.get(url)response.raise_for_status() # Comprovem que la descàrrega ha anat bé
# 2 Guarda el fitxer dins de sdf_files/os.makedirs("sdf_files", exist_ok=True)file_path = "sdf_files/serotonin.sdf"with open(file_path, "w") as f: f.write(response.text)
# 3 Mostra per pantalla les primeres línies del fitxerwith open(file_path) as f: for i in range(5): # Mostrem només 5 línies linia = f.readline() if not linia: break print(linia.strip())
# Aquesta línia informa que hi ha 25 àtoms i 26 enllaços.# 25 26 0 0 0 0 0 0 0 0999 V2000
# 4 Llegeix la molècula amb RDKitmol = Chem.MolFromMolFile(file_path)
# 5 Mostra la fórmula molecular i la notació SMILESformula = rdMolDescriptors.CalcMolFormula(mol)smiles = Chem.MolToSmiles(mol)print(f"\nFórmula molecular: {formula}")print(f"SMILES: {smiles}")
# 6 Guarda una imatge 2D de la molèculaimg = Draw.MolToImage(mol, size=(300, 300))img.save("sdf_files/serotonin.png")print("\n🧪 Imatge guardada com sdf_files/serotonin.png")Escriure fitxers sdf amb RDKIT
Amb RDKit pots fer el procés invers, crear una mol·lecula a partir de la seva representació SMILES.
Anem a veure com crear fitxers .mol i .sdf de la Aspirina (amb SMILES es representa: CC(=O)Oc1ccccc1C(=O)).
from rdkit import Chemfrom rdkit.Chem import AllChemfrom rdkit.Chem import SDWriter
# SMILES i Molsmiles = 'CC(=O)Oc1ccccc1C(=O)O'mol = Chem.MolFromSmiles(smiles)
# Afegir nom a la primera línia del fitxer.mol.SetProp("_Name", "Aspirina")
# Recomanable: Afegir hidrogens explícits que SMILES omet.mol = Chem.AddHs(mol)
# Generar coordenades 3DAllChem.EmbedMolecule(mol, AllChem.ETKDG())
# Guardar a .molwith open("aspirina.mol", "w") as f: f.write(Chem.MolToMolBlock(mol))
# Guardar a .sdfw = SDWriter("aspirina.sdf")w.write(mol)w.close()En realitat, és molt més habitual
Avançat.
RDKit dóna molt més de sí, et permet editar tant la estructura de la mol·lecula (fórmula, enllaços…) com la seva representació (canviar els colors, pintar els àtoms que ens poden interessar més…)
Cobrir-los excedeix del propòsit d’aquesta guia. En part perquè hi ha programari que pot realitzar encara millor aquestes operacions, per exemple PyMol.
Qui tingui interès que consulti: