SMILES és una notació que defineix la estructura de les mol·lècules amb l'ús de cadenes de caràcters ASCII; per guardar-les de forma òptima en sistemes informàtics.
On this page
Introducció
En els vostres estudis de química, començats als primers anys, heu trobat moltes maneres de representar els productes químics, i aquí n’enumerem algunes.
Per exemple,
| Nom trivial | Aspirina |
| Nom sistemàtic | àcid 2-acetiloxibenzoic |
| Fòrmula química | C₉H₈O₄ |
| Fòrmula esquel·lètica | CC(=O)OC1=CC=CC=C1C(=O)O |
| Diagrama | Dibuix |
Com emmagatzemar mol·lecules en sistemes informátics ?
Aquestes formes són molt útils pels professionals de la química.
Ara bé, els reptes de la quimioinformàtica són: guardar, representar i visualitzar els compostos i mol·lècules de forma eficient i còmode tant a nivell químic com informàtic.
Inicialment, podem pensar que el nom sistemàtic (àcid 2-acetiloxibenzoic) o la fórmula química (C9H8O4) ens poden servir per guardar tota la informació d’una substància, però no sempre és així.
Hi pot haver fórmules duplicades, per exemple, tant l’etanol com dimetilèter tenen la fórmula C₂H₆O, però un et fa ballar (alcohol) i l’altre… explotar (èter).
També podriem pensar que per compensar aquesta informació que manca (els enllaços) podem usar la fórmula esquel·lètica.
Afortunadament la IUPAC (Unió Internacional de Química Pura i Aplicada) acorda nomenclatures internacionals de noms químics que són generalment sistemàtics però flexibles. Permeten l’ús de certs noms trivials ben establerts.
Com que els noms IUPAC sistemàtics es fan segons regles formalitzades, en principi podrien ser utilitzats tant per humans com per ordinadors.
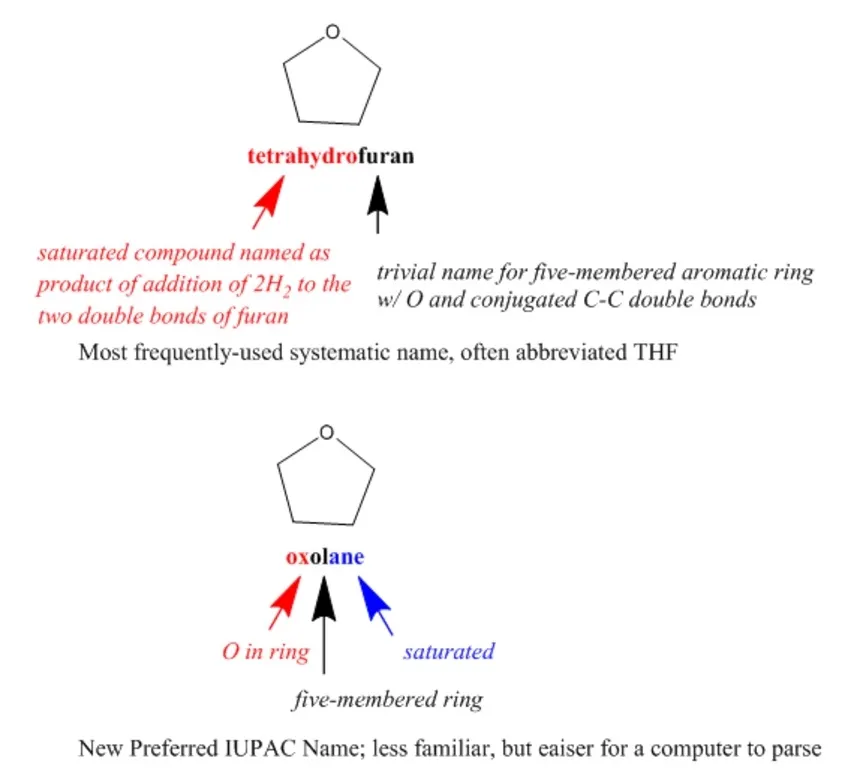
Tanmateix, els noms IUPAC solen ser força difícils de llegir per als químics, i molt menys d’escriure, i les regles no són canòniques, la qual cosa dóna lloc a nombroses opcions diferents per anomenar cada compost.
Amb l’arribada de la quimioinformàtica als anys 70 i 80, es va veure la necessitat de representar molècules d’una forma que fos fàcilment llegible i manipulable per ordinador.
Les primeres solucions van ser les taules de connexions, que descriuen de manera explícita quins àtoms estan units entre si i amb quin tipus d’enllaç.
Els formats de text de taules de connexions encara s’utilitzen força avui en dia (en formats com .mol o .sdf), i els estudiarem més endavant.
Aquestes taules, tot i ser eficients i precises, ocupen força espai i poden resultar lentes de processar quan es treballa amb milers de molècules.
Per això, van sorgir sistemes més compactes, com les notacions lineals, pensades per codificar estructures de forma seqüencial dins de fitxers de text, ocupant menys espai i sent més ràpides de llegir, comparar i transmetre.
Les representacions de notació lineal tenen avantatges clau per a la velocitat i l’automatització, especialment per a manipular un gran nombre d’estructures (per exemple, cercant una gran base de dades).
Per això, les notació de línies es podria considerar com una nomenclatura per a ordinadors, ja que com una taula de connexió, un ordinador pot “llegir” una notació de línies i desenvolupar una molècula de la mateixa manera que un humà pot llegir la nomenclatura IUPAC i generar la molècula. Les notacions de línies són llegibles tant per màquines com per humans.
Actualment, les notacions lineals més utilitzades són el sistema simplificat d’entrada de línies d’entrada molecular (SMILES) i l’identificador químic de la IUPAC (InChI).
En aquesta sessió ens centrarem en la notació SMILES.
Notació SMILES
Les cadenes de SMILES (Simplified Molecular Input Line Entry Specification) són molt útils per ser introduïdes en programes especialitzats i representar molècules sense la necessitat de dibuixar la molècula manualment; és per això que les usare
Hi ha regles específiques sobre com representar àtoms, enllaços, aromaticitat, anells i ramificacions, etc.
Per exemple, els enllaços simples no cal representar-los, però els enllaços dobles es poden representar utilitzant el signe =.
Els àtoms d’hidrogen normalment s’ignoren.
Exemples:
CH2O (Etè) es representa com C=O.
O=C=0 representa el diòxid de Carboni.
O és per representar l’aigua (que la seva fórmula és H2O, però com hem dit els àtoms d’Hidrògen no compten)
Aquí tens una taula esquemàtica de com es relaciona la fórmula química amb la notació SMILES.
| Molècula | SMILES || -------- | ------ || CH₄ | C || NH₃ | N || H₂O | O || CO₂ | O=C=O |En aquesta taula tens uns quants exemples més, de compostos més complexes:
| Compost | SMILES | Notes || ------------------------ | ---------------------------- | ---------------------------------------------------------- || Etanol | `CCO` | Alcohol comú, fàcil de reconèixer. || Glucosa | `C(C1C(C(C(C(O1)O)O)O)O)O` | Molècula de sucre simple, mostra molts OH. || Cafeïna | `Cn1cnc2c1c(=O)n(c(=O)n2C)C` | Molècula amb anells aromàtics i N, típica de bioquímica. || Àcid acètic | `CC(=O)O` | Molècula àcida simple, útil per calcular pKa en exercicis. || Propanona (acetona) | `CC(=O)C` | Molècula simple amb enllaç doble C=O. || Metanamina (methylamine) | `CN` | Mostra un grup amino primari. |OpenSmiles és una versió del llenguatge SMILES per a química patrocinada per la comunitat i basada en estàndards oberts.
Per a consultar més exemples de la notació SMILES consulta a Daylight - Smiles i també a Wikipedia - SMILES.
Taller SMILES i RDKIT
Entorn de treball
Crea un projecte smiles i importa la llibreria RDKit:
uv init smilescd smilesuv add rdkitRDKit és una llibreria de Python molt utilitzada en quimioinformàtica, i que l’equip de desenvolupadors/es actualitza molt sovint.
Tutorials del blog oficial d’RDKit
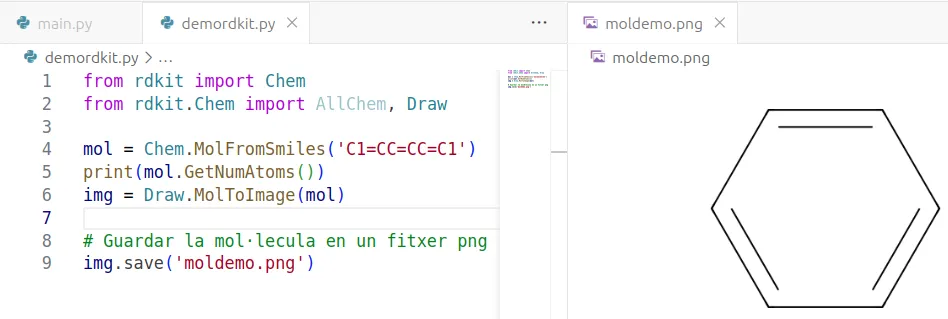
A continuació tens un exemple de codi que usa la biblioteca RDKit per a representar en 2D el Benzè.
Quan crees projectes de software la imatge no es visualitza directament, has de guardar-la en un fitxer png.
from rdkit import Chemfrom rdkit.Chem import AllChem, Draw
mol = Chem.MolFromSmiles('C1=CC=CC=C1')print(mol.GetNumAtoms())img = Draw.MolToImage(mol)
# Si vols guardar la mol·lecula en un fitxer pngimg.save('moldemo.png')El resultat és el següent:
Visualització amb ipynb
Si únicament vols provar si la mol·lecula es veu pots usar el format pynb, que crea JuPyteR Notebooks.
Aquest és molt popular per a científics, ja que no requereix coneixements avançats d’informàtica per arrencar-lo, executes el bloc de codi, esperes i veus els resultats en local o online amb serveis com Google Collab (fitxer exemples RDKit).
De fet, Jupyter és un acrònim de Julia, Python i R, llenguatges típics en el Data Science.
T’interessa saber que existeix el format ipynb per a no tenir errors quan traslladis el codi d’articles online o papers als teus projectes de desenvolupament d’aplicacions, que és el que realment aporta valor afegit a la bioinformàtica.
El mateix codi d’abans amb ipynb quedaria:
from rdkit import Chemfrom rdkit.Chem import AllChem, Draw
mol = Chem.MolFromSmiles('C1=CC=CC=C1')print(mol.GetNumAtoms())
# Mostra com queda la mol·lecula al notebook.Draw.MolToImage(mol)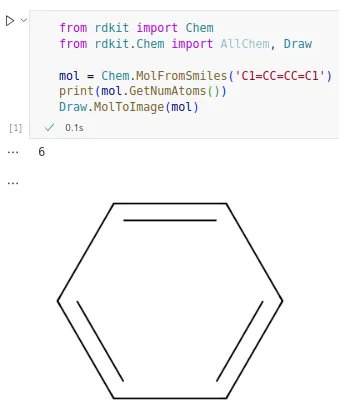
Recorda que el teu IDE et pot demanar baixar-te extensions.
Visualització enllaços
Els àtoms estan representats per l’abreviatura estàndard de cada element químic, entre claudàtors, com ara [Au] per a l’or.
Els claudàtors es poden ometre per a “subconjunts orgànics” de B, C, N, O, P, S, F, Cl, Br, i I.
Tots els altres elements han d’estar tancats entre claudàtors.
Si s’ometen els claudàtors, s’assumeix el nombre propi d’àtoms d’hidrogen implícits; per exemple, la cadena SMILES per a l’aigua és simplement O.
from rdkit import Chemfrom rdkit.Chem import AllChem, Draw
mol = Chem.MolFromSmiles("O")Draw.MolToImage(mol)
img.save('mol-h2o.png')Massa atòmica total
Com ja sabeu, la massa atòmica d’un compost és la suma de la massa de tots els elements que té.
Però RDKit ens facilita la feina recorrent i sumant la massa de tots els àtoms que té 🤓.
from rdkit import Chemfrom rdkit.Chem import AllChem, Draw
# Afegim explicitament els Hidrògens, altrament la massa serà incorrecte.mol = Chem.AddHs(Chem.MolFromSmiles("O"))
compound_mass: int = 0for atom in mol.GetAtoms(): print(atom.GetSymbol()) print(atom.GetAtomicNum()) compound_mass += atom.GetMass() # print(atom.GetMass())
print(f"Massa total del compost: {compound_mass:.4f}")L’aigua és H2O, per tant, pots ometre el H2 perquè està implicit.
Fixa’t, però, que per calcular la massa atòmica hem de tenir en compte les H pel càlcul, i per això usem el mètode auxiliar Chem.AddHs(Chem.MolFromSmiles("O")) per a que ens doni la mol·lecula amb els àtoms d’Hidrògen.
Un àtom que porta una o diverses càrregues elèctriques s’ha de tancar entre claudàtors (sigui quin sigui), seguit del símbol H si està enllaçat a un o diversos àtoms d’hidrogen (aquests van seguits del seu nombre, excepte si només n’hi ha un: NH4 per a l’amoni), després del signe ’+’ per a una càrrega positiva, o del signe ’-’ per a una càrrega negativa.
El nombre de càrregues s’especifica després del signe (excepte si només n’hi ha una); tanmateix, és possible escriure el signe de la/les càrrega/es tantes vegades com càrregues té l’ió: en lloc de “Ti+4”, es pot escriure perfectament “Ti++++” (Titani IV, Ti4+). Així, l’anió hidròxid es representa per [OH-], el catió oxoni per [OH3+], i el catió cobalt III (Co3+) per [Co+3] o per [Co+++].
Obtenir fórmula química.
Per obtenir la fórmula a partir del format Smiles, és tan fàcil com usar la funció CalcMolFormula 🤓:
from rdkit import Chemfrom rdkit.Chem.rdMolDescriptors import CalcMolFormula
mol = Chem.MolFromSmiles("O")# Mostrar la fórmula.formula = CalcMolFormula(mol)print(f"Fórmula química: {formula}")Resultat:
Fórmula química: H2ONombre d’àtoms i enllaços
from rdkit import Chemfrom rdkit.Chem import Descriptors, rdMolDescriptors
mol = Chem.AddHs(Chem.MolFromSmiles("O"))
print("Fórmula:", rdMolDescriptors.CalcMolFormula(mol))print("Massa molecular:", Descriptors.MolWt(mol))print("Massa exacta:", rdMolDescriptors.CalcExactMolWt(mol))print("Nombre d’àtoms:", mol.GetNumAtoms())print("Enllaços:", mol.GetNumBonds())Resultat:
Fórmula: H2OMassa molecular: 18.015Massa exacta: 18.010564684Nombre d’àtoms: 3Enllaços: 2Informació de cada àtom
Per obtenir informació d’un àtom, o de cada àtom que forma una mol·lecula, podem obrir un bucle per recórre’ls tots.
Així, podrem obtenir el símbol, la massa, el número atòmic, fins i tot la valència.
from rdkit import Chem
mol = Chem.AddHs(Chem.MolFromSmiles("O=C=O")) # diòxid carboni
for atom in mol.GetAtoms(): symbol = atom.GetSymbol() val_total = atom.GetTotalValence() print(f"{symbol}: valència={val_total}")Resultat:
O: valència=2C: valència=4O: valència=2Exercicis RDKit
Per familiaritzar-te amb SMILES i RDKit prova el codi amb un compost que et sembli interessants.
Concretament, agafa un compost i crea un programa que obtingui 🤔🤔:
- Visualització enllaços
- Massa atòmica
- Fórmula química
from rdkit import Chemfrom rdkit.Chem import Draw, Descriptors, rdMolDescriptors
mol_exemple = "OC(C1C(C(C(C(O1)O)O)O)O)O"print(f"Mostrant el compost: {mol_exemple}")
# Guardar la mol·lecula en una imatge.mol = Chem.MolFromSmiles(mol_exemple)img = Draw.MolToImage(mol)img.save('mol_exemple.png')
# Mostrar informació de la mol·leculamol_Hs = Chem.AddHs(Chem.MolFromSmiles(mol_exemple))
print("Fórmula:", rdMolDescriptors.CalcMolFormula(mol_Hs))print("Massa molecular:", Descriptors.MolWt(mol_Hs))print("Massa exacta:", rdMolDescriptors.CalcExactMolWt(mol_Hs))print("Nombre d’àtoms:", mol_Hs.GetNumAtoms())print("Enllaços:", mol_Hs.GetNumBonds())Referències
- Representing Small Molecules on Computers
- Getting Started with the RDKit in Python
- Tutorial to SMILES and canonical SMILES explained with examples
- Molecular Deep Learning using DeepChem
- React MolViewer
TODO: Molecular Fingerprinting
Molecular fingerprints are a way to encode a molecule’s structure directly as a bit vector. There are several algorithms but a commonly used one is the ECFP4 notation that captures both the atom’s properties and the properties of neighboring atoms within a diameter of 4.
Then a hashing function is used to convert this identifier into a fixed-size bit vector [7].
A typical application of this representation is to find molecular similarity.
Graph
Another common representation of a molecule is as a Graph.
E.g., Adjacency Matrix. If we imagine molecules as graphs, then a node represents an atom, the edge represents the bond between the two atoms. Graphical representations can be more advantageous especially if spatial differences are important as it can preserve this data as opposed to a linear data model like SMILES.