Fitxers
Llegir i editar fitxers de text amb Python és molt senzill, fins i tot si venen comprimits. També veurem com controlar les possibles excepcions durant l'accés als fitxers.
Llegir, crear i editar fitxers de text de tot tipus (text pla, binaris, CSV, JSON, HTML… ) amb Python és ràpid i molt senzill.
Conceptes previs.
Section titled “Conceptes previs.”Abans de començar a treballar amb fitxers a Python, és important entendre com els sistemes operatius moderns gestionen els fitxers.
En el seu nucli, un fitxer és un conjunt contigu de bytes utilitzats per emmagatzemar dades. Aquestes dades s’organitzen en un format específic i poden ser qualsevol cosa tan simple com un fitxer de text o tan complicat com un programa executable.
Nosaltres ens centrarem en el primer cas, fixers en format de text pla.
Els fitxers de la majoria dels sistemes de fitxers moderns es componen de tres parts principals:
- Capçalera: metadades sobre el contingut del fitxer (nom del fitxer, mida, tipus, etc.)
- Dades: contingut del fitxer tal com l’ha escrit el creador o editor
- Final del fitxer (EOF): caràcter especial que indica el final del fitxer.
Rutes (Paths)
Section titled “Rutes (Paths)”Quan accediu a un fitxer en un sistema operatiu, cal una ruta de fitxer.
La ruta del fitxer és una cadena que representa la ubicació d’un fitxer. Està dividit en tres grans parts:
- Ruta de la carpeta: la ubicació de la carpeta de fitxers al sistema de fitxers on les carpetes posteriors estan separades per una barra inclinada
/(Unix,Linux i MacOS) o una barra invertida\(Windows) - Nom del fitxer: el nom real del fitxer
- Extensió: el final de la ruta del fitxer amb un punt ( .) que s’utilitza per indicar el tipus de fitxer
Anem a analitzar un cas molt habitual; imaginem que volem obrir el fitxer animals.csv des del programa read_file.py.
/│├── python_files/ ← working directory| │| ├── data/ ← Current working directory (cwd)| │ └── animals.csv| │| └── read_file.py ← Accessing this fileLa ruta del fitxer correcta per tractar-lo en aquest cas, suposant que estem a Linux, seria:
file_path = "data/animals.csv"És molt important l’ús de rutes de fitxers en aquest format, que es tracta d’una ruta relativa.
Cal evitar sempre les rutes absolutes (per exemple: C:\Users\usuari1\file.py) o /home/user1 perquè només funcionen dins la màquina local i poden conduir a forats de seguretat.
Lectura de fitxers bàsica.
Section titled “Lectura de fitxers bàsica.”Per tal d’obrir un fitxer de text per treballar-hi; només cal usar la funció integrada open(), que només necessita la ruta del fitxer.
També és important que sabeu que la paraula reservada with ens permet obrir el fitxer i tancar-lo automàticament.
⚠Avís: Recomanem usar `with open() per tal d’assegurar-vos que el fitxer es tanqui correctament i així evitar problemes!⚠
file_name = 'demo.txt'with open(file_name, mode='r') as file: print(file.read())
print(f"Fitxer {file_name} llegit correctament.")Modes d’accés als fitxers: lectura i escriptura.
Section titled “Modes d’accés als fitxers: lectura i escriptura.”Fixem-nos de nou el bloc de codi que obre el fitxer, llegeix i mostra el seu contingut i el tanca quan ha acabat:
with open(file_name, mode='r') as file: print(file.read())Apart de tenir el paràmetre necessari del file_name, informem el segon paràmetre, el mode, que pot tenir aquests valors:
- ‘r’ Obert per llegir
read(per defecte) - ‘w’ Obriu per escriure
write, truncant (sobreescrivint) el fitxer primer si existeix. - ‘a’ Obriu per escriure
append, si el fitxer existeix posa el contingut nou al final. - ‘rb’o’wb’ Obrir en mode binari (llegir/escriptura mitjançant dades de bytes)
Per exemple, si volem afegir una nova linia al final d’un fitxer existent, és tan senzill com usar aquest codi:
with open('data/demo.txt', 'a') as appender: appender.write("\nHave a nice day!")Encoding: Codificació de caràcters i salts de línia
Section titled “Encoding: Codificació de caràcters i salts de línia”Tingueu en compte que el format de codificació de caràcters del fitxer ha de ser l’adient per evitar errors.
Per exemple, si s’ha creat un fitxer amb la codificació UTF-8 (Unicode), amb millions de caràcters i intenteu analitzar-lo amb la codificació ASCII (128 caràcters), si hi ha un caràcter que es troba fora d’aquests 128 valors, es generarà un error.
També heu de tenir en compte Windows utilitza els caràcters \r\n per indicar una línia nova, mentre que Unix, Linux i les versions més noves de Mac utilitzen només el caràcter \n. Això pot provocar algunes complicacions quan esteu processant fitxers en un sistema operatiu diferent de l’origen del fitxer.
Anem a veure un exemple senzill.
Si a demo.txt tenim aquest contingut:
Com esteu?Espero que molt bé!Hem de tenir en compte un altre paràmetre: encoding="UTF-8"
with open('demo.txt', mode='r', encoding="UTF-8") as reader: # Further file processing goes here # Read & print the entire file print(reader.read())
print("Fitxer demo.txt llegit correctament.")Ja que sinó, el programa no interpretarà els accents ni altres caràcters é :
Com esteu?Espero que molt bé!Fitxer demo.txt llegit correctament.Activitat bàsica.
Section titled “Activitat bàsica.”0.- Abans de començar la activitat, crea els següents directoris i crea el fitxer oferta1.txt. Ho pots fer amb entorn gràfic o per terminal.
/│├── python_files/ ← working directory| │| ├── data/ ← Current working directory (cwd)| │ └── oferta1.txt| │| └── read_file.py ← Accessing this fileEl text del fitxer oferta1.txt ha de ser:
PythonPandasMatplotlibFastAPI3dJSReactMariaDB1.- Fes un programa que obri el fitxer, el llegeixi i mostri tot el seu contingut per pantalla.
{% sol %}
with open('data/oferta1.txt', 'r') as reader: print(reader.read()){% endsol %}
2.- Crea un altre programa que obri el fitxer i a la última línia escrigui el següent text:
Oferta revisada.{% sol %}
with open('data/demo.txt', 'a') as appender: appender.write("\nOferta revisada."){% endsol %}
Tractament excepcions
Section titled “Tractament excepcions”Les excepcions a Python són una eina molt potent que la gran majoria de llenguatges de programació moderns tenen. Es tracta d’una manera de controlar el comportament d’un programa quan es produeix un error.
Això és molt important ja que sinó tractem aquest error, el programa es pararà, i això no és una opció vàlida quan publiquem la nostra aplicació.
Tenim el llistat d’excepcions al web oficial Pyhton.
Per exemple, suposem que creem un programa per dividir 2 números que els usuaris ens enviin. Contemplem dos possibles excepcions:
- Que el divisor (segon paràmetre) sigui 0.
- O que l’usuari envii text en comptes de números.
Anem a veure com realitzar correctament la divisió i tractar aquestes excepcions:
Si encara no el tens, crea un projecte amb poetry:
poetry new demo-files --name appObre’l amb VSCode:
code demo-filesAfegeix la dependència pytest:
poetry demo-filesI activa :
poetry demo-filesTot llest 😉 ? Genial, ja podem crear els tests i la funció:
Per tractar les excepcions el que fa Python és descomposar el codi en 3 blocs:
- try és el tros de codi que preveiem que pot donar excepcions.
- except és un tros de codi que volem que s’executi si tenim alguna excepció. En aquest cas tenim 2 possibles excepcions:
ZeroDivisionErroriValueError. La paraularaiseserveix per enviar la excepció per a què el programa principal o un altre funció la pugui tractar. - finally és un tros de codi que s’executarà sempre, tant si tot ha funcionat com si hi ha hagut algun error. En aquest cas no el necessitem.
Per a què funcioni el bloc try hi ha d’haver o un except o un finally (sinó, no té sentit crear un try).
def dividir(a, b): """Realitza la divisió de a entre b i captura errors comuns.""" try: # Intenta convertir a i b a floats per evitar strings a = float(a) b = float(b) resultat = a / b print(f"El resultat de {a} / {b} és: {resultat}") return resultat except ZeroDivisionError: raise ZeroDivisionError("No es pot dividir per zero!") except ValueError: raise ValueError("Els valors han de ser números (no strings).")Si provem d’executar la divisió en un cas excepcional:
print(dividir("a","b"))Veurem que ha capturat la excepció i la ha transformat en el missatge que volem transmetre a l’usuari.
Traceback (most recent call last): File "/mnt/c/Users/USUARI/m14/s6/demo1/app/dividir.py", line 15, in <module> print(dividir("a","b")) File "/mnt/c/Users/USUARI/m14/s6/demo1/app/dividir.py", line 13, in dividir raise ValueError("Els valors han de ser números (no strings).")ValueError: Els valors han de ser números (no strings).El codi que executa tests de tots els casos habituals i exepcionals el tens aquí.
import pytest# from dividir import dividir
def test_divisio_correcta(): assert dividir(10, 2) == 5
def test_divisio_per_zero(): with pytest.raises(ZeroDivisionError, match="No es pot dividir per zero!"): dividir(10, 0)
def test_divisio_string(): with pytest.raises(ValueError): dividir("a", 5) with pytest.raises(ValueError): dividir(5, "b") with pytest.raises(ValueError): dividir("a", "b")El que canvia en els casos excepcionals és que posem el mètode dins d’aquest bloc:
with pytest.raises(ZeroDivisionError, match="No es pot dividir per zero!"):Per a capturar que la excepció que volem que apareixi a l’usuari és la que desitgem.
Arrenca els tests:
pytestResultat:
collected 3 items
demo1/tests/dividir_test.py ... [100%]
=============================== 3 passed in 0.32s ===============================Ja sabem com crear funcions més robustes i tolerants a errades 😀!
Tractament d’excepcions en fitxers.
Section titled “Tractament d’excepcions en fitxers.”Ara que ja hem repassat com funcionen les excepcions en Python, ja podem aplicar tot el que hem vist per tractar-les en l’acccés a fitxers.
Recordem que Python descomposa el codi que pot donar exepcions en en 3 blocs, i un d’opcional:
- try és el tros de codi que preveiem que pot donar excepcions.
- except el trps de codi que tracta excepció. En el cas de l’accés a fitxers tenim aquests casos:
ZeroDivisionErroriValueError. - finally és un tros de codi que s’executarà sempre, tant si tot ha funcionat com si hi ha hagut algun error. En aquest cas no el necessitem.
- else si després d’executar el try volem executar més operacions només si ha funcionat podem usar l’else.
Per a què funcioni el bloc try hi ha d’haver o un except o un finally (sinó, no té sentit crear un try).
En resum:
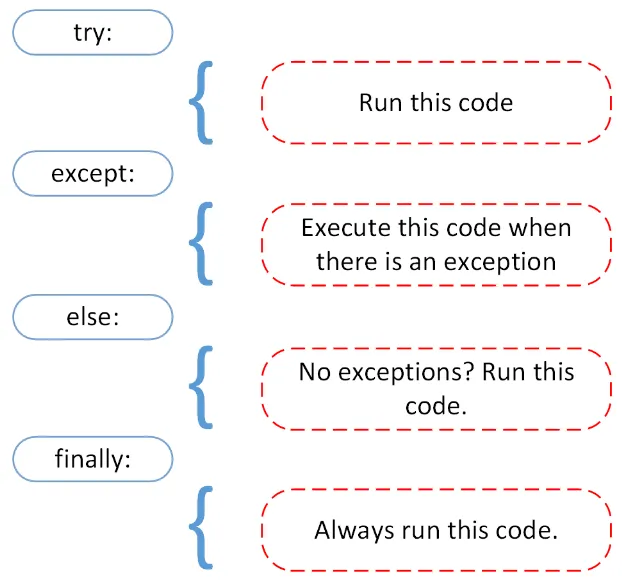
Anem a veure com funciona a la pràctica.
Si volem llegir un fitxer amb el nom de 6 bioinformàtiques i els descobriments que han aportat a la humanitat; i guardar-les en una llista.
bioinformatiques1.csv
Nom,Descobriment Famos,DatesAnna Smith,Desenvolupament d'algoritmes per anàlisi de seqüències d'ADN,1960-Maria Garcia,Creació de bases de dades genòmiques,1955-Laura Johnson,Innovacions en anàlisi de xarxes gèniques,1965-Patricia Brown,Desenvolupament de models predictius en bioinformàtica,1970-Linda Davis,Contribucions a l'anàlisi de dades d'expressió gènica,1945-2010Barbara Martinez,Investigació en genòmica del càncer,1968-Les possibles excepcions que preveiem seran:
- FileNotFoundError —> No trobem el fitxer.
- OSError —> Que l’usuari no té permisos per accedir-hi.
- Exception —> Aquesta excepció captura qualsevol altre error que pugui sorgir. És bona pràctica incloure-la per poder depurar i cercar solucions.
En els 2 últims casos el que fem és mostrar la traça de l’excepció complerta, per exemple: except Exception as e
El codi final serà:
fitxer = 'bioinformatiques1.csv'# Llista per guardar les línies del fitxerlinies = []
try: with open(fitxer, mode='r', encoding='utf-8') as file: # Ometre la primera fila (encapçalats) next(file) for line in file: try: # El separador de cada dada del fitxer és ',' nom, descobriment, dates = line.strip().split(',') linies.append((nom, descobriment, dates)) except ValueError as e: print(f"Error al processar la línia: {line.strip()}. Error: {e}") exit() #Forcem la sortida del programa.
except FileNotFoundError: print(f"El fitxer {fitxer} no s'ha trobat.")except OSError as e: print(f"S'ha produït un error del sistema en intentar accedir al fitxer: {e}")except Exception as e: print(f"S'ha produït un error inesperat: {e}")else: print(f"Hem aconseguit llegir tot el fitxer correctament!") print(linies[0])finally: print("Gràcies per confiar en el nostre programari.")Provem de llegir el fitxer; primer amb el nom exacte.
file_path = Path('bioinformatiques1.csv')app-py3.10miquel@LAPTOP-NBA651HM:/mnt/c/Users/USUARI/m14/s6$ python files-bio-exceptions.pyHem aconseguit llegir tot el fitxer correctament!('Anna Smith', "Desenvolupament d'algoritmes per anàlisi de seqüències d'ADN", '1960-')Gràcies per confiar en el nostre programari.Si ha funcionat, ara provarem de forçar una excepció per veure si es captura correctament: canviem el nom del fitxer a llegir per un que no existeixi i executem de nou el codi.
file_path = Path('bioinformatics-none.csv')Com que no tenim el fitxer, ens surt la excepció que hem programat. Així podem aportar una millor experiència als/les usuaris/es de l’aplicació.
app-py3.10miquel@LAPTOP-NBA651HM:/mnt/c/Users/USUARI/m14/s6$ python files-bio-exceptions.pyEl fitxer bioinformatiques1123.csv no s'ha trobat.Gràcies per confiar en el nostre programari.Una altra excepció que podem tenir és que alguna de les línies no tingui el separador correcte, que en el nostre cas és la ,. Podem provar a veure si ho detecta adequadament, canviant la , per uns espais :
Nom,Descobriment Famos,DatesAnna Smith Desenvolupament d'algoritmes per anàlisi de seqüències d'ADN,1960-Maria Garcia,Creació de bases de dades genòmiques,1955-Si executem el codi ens surt l’error que hem esperat. Li hem forçat de sortir del programa amb la instrucció exit(), però en programes en producció no hauriem de usar exit()
app-py3.10miquel@LAPTOP-NBA651HM:/mnt/c/Users/USUARI/m14/s6$ python files-bio-exceptions.pyError al processar la línia: Anna SmithxdDesenvolupament d'algoritmes per anàlisi de seqüències d'ADN,1960-. Error: not enough values to unpack (expected 3, got 2)Gràcies per confiar en el nostre programari.Lectura / Escriptura de diversos fitxers.
Section titled “Lectura / Escriptura de diversos fitxers.”Suposem que volem llegir dos fitxer de text que contenen dones referents en la bioinformàtica i els volem guardar en una llista per a processar-los millor.
Aquests són els fitxers:
bioinformatiques1.csv
Nom,Descobriment Famos,DatesAnna Smith,Desenvolupament d'algoritmes per anàlisi de seqüències d'ADN,1960-Maria Garcia,Creació de bases de dades genòmiques,1955-Laura Johnson,Innovacions en anàlisi de xarxes gèniques,1965-Patricia Brown,Desenvolupament de models predictius en bioinformàtica,1970-Linda Davis,Contribucions a l'anàlisi de dades d'expressió gènica,1945-2010Barbara Martinez,Investigació en genòmica del càncer,1968-bioinformatiques2.csv
Elizabeth Rodriguez,Desenvolupament d'eines bioinformàtiques per l'epigenètica,1973-Jennifer Hernandez,Estudis sobre variació genètica humana,1980-Susan Lopez,Desenvolupament de programari per anàlisi de proteïnes,1962-Margaret Gonzalez,Recerca en metagenòmica,1958-Dorothy Wilson,Estudis sobre l'evolució molecular,1975-Sarah Anderson,Desenvolupament de metodologies per seqüenciació de nova generació,1983-Jessica Thomas,Investigació en biologia computacional estructural,1972-Karen Taylor,Desenvolupament de bases de dades de proteïnes,1960-Nancy Moore,Innovacions en bioinformàtica de sistemes,1971-Betty Jackson,Recerca en biologia de xarxes,1952-Helen White,Desenvolupament d'eines per anàlisi de RNA-seq,1985-Sandra Harris,Estudis sobre interaccions proteïna-proteïna,1976-Donna Martin,Recerca en farmacogenòmica,1967-Carol Thompson,Desenvolupament de metodologies per l'anàlisi de dades de microarrays,1970-Per poder llegir els 2 fitxers (o més) d’un sol cop podem usar aquest codi:
from pathlib import Path
# Obtenir tots els fitxers CSV del directori actualfitxers = [Path('bioinformatiques1.csv'), Path('bioinformatiques2.csv')]# Alternativa:#fitxers = list(Path('.').glob('*.csv'))
# Llista per acumular les dadeslinies = []
for fitxer in fitxers: try: with fitxer.open(mode='r', encoding='utf-8') as file: # Ometre la primera fila (encapçalats) next(file) for line in file: try: nom, descobriment, dates = line.strip().split(',') linies.append((nom, descobriment, dates)) except ValueError as e: print(f"Error al processar la línia: {line.strip()}. Error: {e}") except FileNotFoundError: print(f"El fitxer {fitxer} no s'ha trobat.") except OSError as e: print(f"S'ha produït un error en intentar accedir al fitxer: {e}") # Aquesta excepció captura qualsevol altre error que pugui sorgir (és bona pràctica per depurar) except Exception as e: print(f"S'ha produït un error inesperat: {e}")
print(linies)Fixem-nos amb les noves linies que hem aplicat:
- Path —> Llibreria que permet agafar un fitxer sense haver-nos de preocupar pels detalls de la ruta, els símbols
/i conté utilitats per llegir fitxers. - for fitxer in fitxers: —> Llegeix tots els fitxers seleccionats (si n’hi ha almenys un)
- for line in file: —> Llegeix linia a linia.
- next —> Salta a llegir la següent fila del fitxer.
- line.strip().split(’,’) -> Ens interessa separar els valors de cada linia separats per comes.
Fitxer dins de diccionari.
Section titled “Fitxer dins de diccionari.”Anteriorment hem vist com n’és de còmode utilitzar els diccionaris per consultar informació agrupada en {% link “/python/data/” %}, és a dir, diccionaris.
És especialment útil per tractar fitxers CSV.
Per exemple, podem guardar en un diccionari els 5 articles de medicina amb la puntuació SJR més alta, que serveix per mesurar l’impacte dels articles científic basat en el número de citacions. També és important l’H-Index.
La Font de dades del CSV és el portal Scimago Journals
És una organització que analitza mitjançant diverses mètriques la qualitat de les publicacions cientìfiques de diversos llocs del món: l’SRJ, l’H-Index, el total absolut d’articles citats, documents publicats durant 1 i 3 anys…
Crea un fitxer anomenat scimagojr-2023.csv amb aquest contingut:
Rank;Sourceid;Title;Type;Issn;SJR;SJR Best Quartile;H index;Total Docs. (2023);Total Docs. (3years);Total Refs.;Total Cites (3years);Citable Docs. (3years);Cites / Doc. (2years);Ref. / Doc.;%Female;Overton;SDG;Country;Region;Publisher;Coverage;Categories;Areas1;28773;"Ca-A Cancer Journal for Clinicians";journal;"15424863, 00079235";106,094;Q1;211;49;124;4844;35427;89;381,89;98,86;43,95;2;35;United States;Northern America;"Wiley-Blackwell";"1950-2023";"Hematology (Q1); Oncology (Q1)";"Medicine"2;12464;"Nature Reviews Cancer";journal;"1474175X, 14741768";26,837;Q1;505;105;304;10805;10951;163;31,23;102,90;44,33;1;59;United Kingdom;Western Europe;"Nature Publishing Group";"2001-2023";"Cancer Research (Q1); Oncology (Q1)";"Biochemistry, Genetics and Molecular Biology; Medicine"3;20425;"Nature Reviews Drug Discovery";journal;"14741784, 14741776";22,399;Q1;391;239;731;8584;13091;153;19,72;35,92;34,15;5;61;United Kingdom;Western Europe;"Nature Publishing Group";"2002-2023";"Drug Discovery (Q1); Medicine (miscellaneous) (Q1); Pharmacology (Q1)";"Medicine; Pharmacology, Toxicology and Pharmaceutics"4;17700156734;"Nature Reviews Clinical Oncology";journal;"17594782, 17594774";21,048;Q1;217;127;400;9888;10807;183;28,36;77,86;38,85;0;56;United Kingdom;Western Europe;"Nature Publishing Group";"2009-2023";"Oncology (Q1)";"Medicine"5;15847;"New England Journal of Medicine";journal;"00284793, 15334406";20,544;Q1;1184;1388;4522;14603;107246;1824;21,69;10,52;38,26;0;576;United States;Northern America;"Massachussetts Medical Society";"1945-2023";"Medicine (miscellaneous) (Q1)";"Medicine"I ara crea el codi bàsic per llegir el fitxer. Aquest s’ajuda de la llibreria predefinida csv.
from csv import DictReader
# How to define a function in python with the word key# the type date after the : is only documentation for Pythondef read_csv_file(csv_file_path: str) -> list[dict]:
with open(csv_file_path, newline='') as csv_file: csv_reader = DictReader(csv_file, delimiter=';') result = [row_dict for row_dict in csv_reader]
return result
csv_file_path: str = "scimagojr-2023.csv"entries: list[dict] = read_csv_file(csv_file_path)print(entries)print(f"There are {num} entries.")Activitat.
Section titled “Activitat.”1.- Insereix el tractament d’excepcions al codi anterior.
{% sol %}
import csv
def read_csv_file(csv_file_path: str) -> list[dict]: """Llegeix un fitxer CSV i retorna una llista de diccionaris amb les seves entrades.""" try: with open(csv_file_path, newline='', encoding='utf-8') as csv_file: csv_reader = csv.DictReader(csv_file, delimiter=';') result = [row_dict for row_dict in csv_reader] except FileNotFoundError: raise FileNotFoundError(f"El fitxer {csv_file_path} no s'ha trobat.") except OSError as e: raise OSError(f"Error d'accés al fitxer: {e}") except csv.Error as e: raise ValueError(f"Error en llegir el fitxer CSV: {e}") else: return result
# Exemple d'úsif __name__ == "__main__": csv_file_path = "scimagojr-2023.csv" try: entries = read_csv_file(csv_file_path) print(entries) print(f"There are {len(entries)} entries.") except Exception as e: print(f"Ha ocorregut un error: {e}"){% endsol %}
2.- Verifica el funcionament correcte del programa amb 2 o 3 mètodes de test amb pytest; tant en casos habituals com en excepcions.
{% sol %}
import pytestfrom scimago import read_csv_file
def test_read_csv_correct(): """Comprova que es llegeix correctament un fitxer CSV.""" csv_file_path = "scimagojr-2023.csv" result = read_csv_file(csv_file_path) assert len(result) > 0 assert "Title" in result[0] # Comprovem que el fitxer té una columna 'Title'
def test_file_not_found(): """Comprova que es llança FileNotFoundError quan el fitxer no existeix.""" with pytest.raises(FileNotFoundError): read_csv_file("no-existeix.csv"){% endsol %}
3.- Crea un codi que serveixi per consultar els articles de UK. La columna a consultar és country i el valor del país United Kingdom.
{% sol %}
if __name__ == "__main__": csv_file_path = "scimagojr-2023.csv" try: entries = read_csv_file(csv_file_path) # Consulta articles del Regne Unit (UK). Columna = `country`, valor = `United Kingdom` uk_entries = [entry for entry in entries if entry.get('country') == 'United Kingdom'] print(f"Nombre d'entrades del Regne Unit: {len(uk_entries)}") for entry in uk_entries: print(entry)
except Exception as e: print(f"Ha ocorregut un error: {e}"){% endsol %}
Opcionalment, tens més exemples de consultes a:
Descomprimir fitxers.
Section titled “Descomprimir fitxers.”Si tenim un fitxer zip amb diversos fitxers i volem descomprimir-los automàticament per treballar amb ells podem seguir aquest codi:
from zipfile import ZipFile, BadZipFilefrom pathlib import Path
# Definim el camí al fitxer ZIP i la carpeta de destízip_file_path = Path("students.zip")destination_folder = Path("students")
try: with ZipFile(zip_file_path, 'r') as zObject: zObject.extractall(path=destination_folder) print("Fitxer ZIP extret amb èxit!")
except FileNotFoundError: print(f"Error: El fitxer '{zip_file_path}' no es troba.")except BadZipFile: print("Error: El fitxer no és un ZIP vàlid.")except Exception as e: print(f"Ha ocorregut un error inesperat: {e}")Per saber com des/comprimir fitxers .tar.gz i altres funcionalitat segueix llegint:
- [https://realpython.com/working-with-files-in-python/#extracting-zip-archives](Blog Real Python - Extracting Zip archives)
Referències consultades:
Section titled “Referències consultades:”El contingut d'aquest lloc web té llicència CC BY-NC-ND 4.0.
©2022-2025 xtec.dev