On this page
Introducció
La reacció en cadena de la polimerasa, o PCR, és una tècnica per a fer moltes còpies d’una determinada regió d’ADN in vitro. La reacció en cadena de la polimerasa (PCR) és una manera eficaç i eficient de copiar petites seqüències específiques d’ADN o ARN i té moltes aplicacions en biologia, medicina i biotecnologia.
La PCR depèn d’una ADN polimerasa termoestable, la Taq polimerasa, i requereix l’ús de parelles d’oligonucleòtids com a iniciadors o primers d’ADN dissenyats específicament per a la regió d’ADN d’interès. En la PCR, la reacció se sotmet repetidament a un cicle de canvis de temperatura que permeten la producció de moltes còpies de la regió blanc.
En general, l’objectiu de la PCR és produir suficient ADN de la regió diana perquè es pugui analitzar o utilitzar d’alguna altra manera. Per exemple, l’ADN amplificat per PCR es pot seqüenciar, visualitzar per electroforesi en gel o clonar en un plasmidi per a altres experiments.
Per això, un dels paràmetres més importants per a tindre èxit en l’amplificació per PCR és el disseny correcte de la parella d’oligonucleòtids / Primers. Perquè si aquests no estan ben dissenyats, la PCR no funcionarà de manera adequada.
Per això, finalment analitzarem les principals variables a tindre en compte a l’hora de dissenyar primers/oligonucleòtids, entre ells:
- Temperatura de fusió.
- Grandària de l’oligonucleòtid.
- Especificitat.
- Complementarietat en la seqüència dels oligonucleòtids.
La Taq polimerasa
Igual que la replicació d’ADN en un organisme, la PCR requereix d’un enzim ADN polimerasa que produeixi noves cadenes d’ADN mitjançant l’ús de les cadenes existents com a motle. L’ADN polimerasa que normalment s’utilitza en la PCR es diu Taq polimerasa, pel bacteri tolerant a la calor de la qual es va aïllar (Thermus aquaticus).
T. aquaticus viu en aigües termals i fonts hidrotermals. El seu ADN polimerasa és molt termoestable i la seva major activitat es presenta prop dels 70°C (temperatura a la qual l’ADN polimerasa d’ésser humà o d’E. coli no funcionaria). La Taq polimerasa és ideal per a la PCR gràcies a aquesta estabilitat tèrmica. Com veurem, la PCR utilitza altes temperatures repetidament per a desnaturalitzar el motle d’ADN o separar les seves cadenes.
Primers per PCR
De la mateixa manera que unes altres ADN polimerases, la Taq polimerasa només pot fer ADN si hi ha un primer, una curta seqüència de nucleòtids que proporciona un punt de partida per a la síntesi d’ADN. En una reacció de PCR, la regió d’ADN que serà copiada, o amplificada, es determina pels primers que la investigadora escolleix.
Els primers per a PCR són trossos curts d’ADN de cadena senzilla, generalment d’uns 20 nucleòtids de longitud. En cada reacció de PCR s’utilitzen dos primers que estan dissenyats per a flanquejar la regió diana (la regió que ha de ser copiada). És a dir, els agreguen seqüències que faran que s’unisquen a cadenes oposades del motle d’ADN només en els extrems de la regió a copiar. Els primers s’uneixen al motle mitjançant complementarietat de bases.
Quan els primers s’uneixen al motle, la polimerasa els estén i la regió que es entre ells es copia.

Tots dos primers apunten “cap a dintre” en unir-se, és a dir, en direcció 5’ a 3’ cap a la regió a copiar. Igual que unes altres ADN polimerases, la Taq polimerasa només pot sintetitzar ADN en direcció 5’ a 3’. En estendre’s els primers, la regió que es troba entre ells es copia.
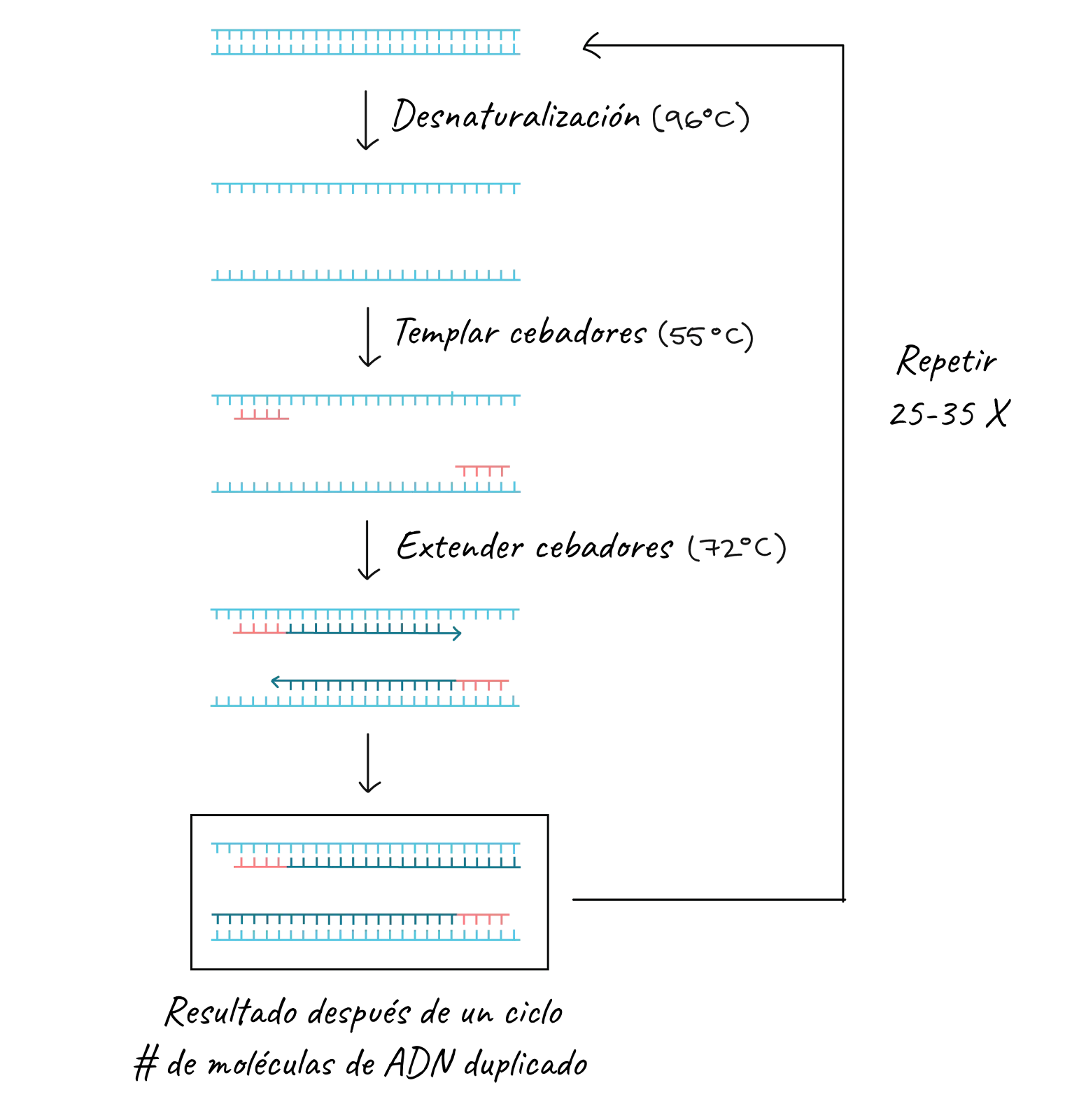
Els passos de la PCR
Com ja hem dit, els ingredients clau per a una reacció de PCR són Taq polimerasa, primers, ADN motle i nucleòtids (els blocs bàsics de l’ADN). Els ingredients es col·loquen en un tub, juntament amb els cofactors que necessiten l’enzim, i se sotmeten a cicles repetits de calfament i refredament que permeten la síntesi de l’ADN.
Els passos bàsics són:
- Desnaturalització (96°C): la reacció es calfa bastant per a separar, o desnaturalitzar, les cadenes d’ADN. Això proporciona els motles de cadena senzilla per al següent pas.
- Temperatura (55 - 65°C): la reacció es refreda perquè els primers puguen unir-se a les seues seqüències complementàries en el motle d’ADN de cadena senzilla.
- Extensió (72°C): la temperatura de la reacció s’eleva perquè la Taq polimerasa estenga els primers i sintetitze així noves cadenes d’ADN.
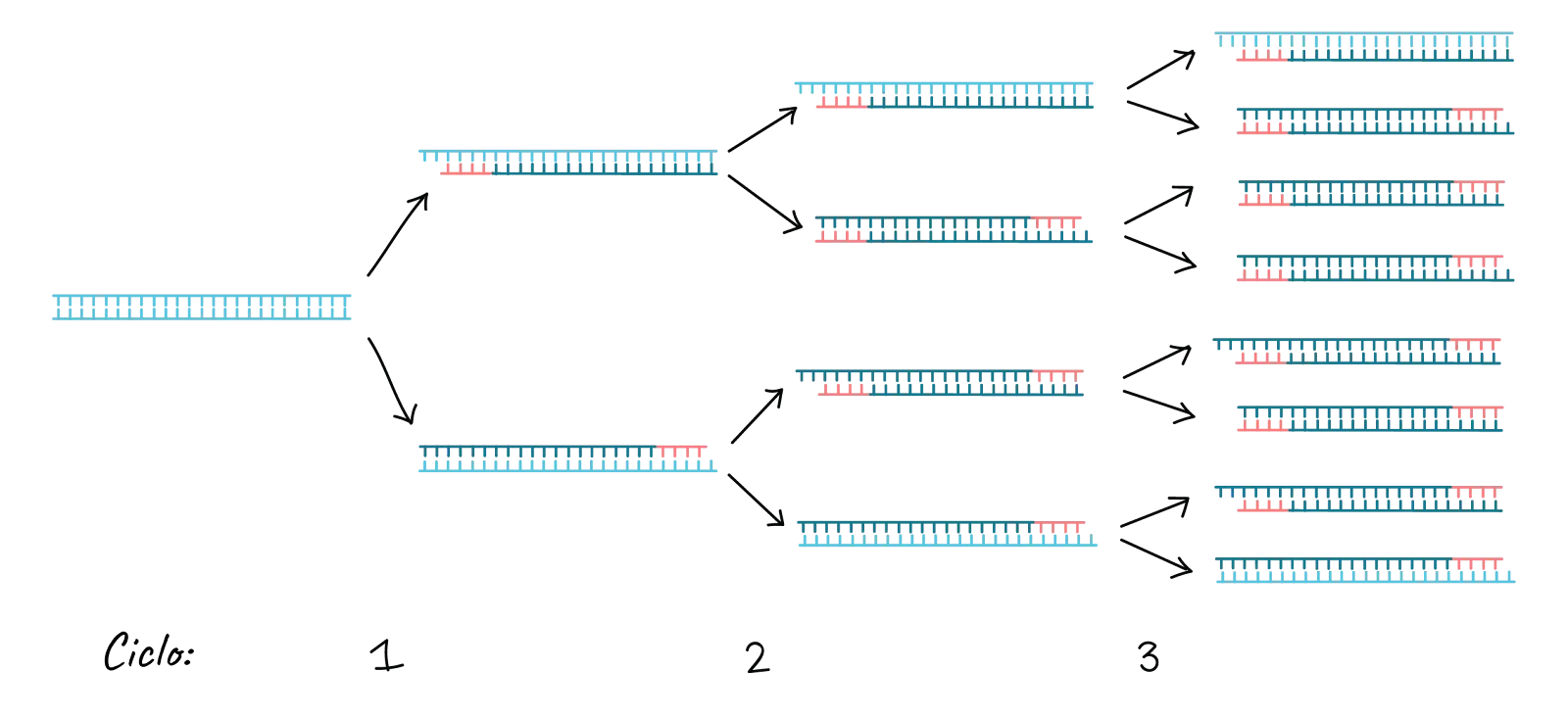
Aquest cicle es repeteix 25-35 vegades en una reacció de PCR típica, que generalment triga 2-4 hores segons la longitud de la regió d’ADN que es copia. Si la reacció és eficient (funciona bé), pot produir milers de milions de còpies a partir d’una o unes quantes còpies de la regió diana. Això és perquè no sols s’usa l’ADN original com a motle en cada cicle. En realitat, el nou ADN que es produeix en una ronda pot servir com a motle en la següent ronda de síntesi d’ADN. Hi ha moltes còpies dels engreixadors i moltes molècules de Taq polimerasa surant en la reacció, per la qual cosa el nombre de molècules d’ADN quasi pot duplicar-se en cada cicle. La següent imatge mostra aquest patró de creixement exponencial:
Principals variables a l’hora de fer una PCR
Temperatura de fusió
La temperatura de fusió (Tm) és la temperatura a la qual la meitat dels dobles brins d’ADN estan desnaturalitzats. És important tindre en compte que en una reacció de PCR hi ha dos oligonucleòtids i tots dos haurien de dissenyar-se de manera que tinguen Tm similars. Si els oligonucleòtids no tenen Tm semblants, l’amplificació serà menys eficient o fins i tot pot no funcionar ja que l’oligonucleòtid amb la Tm més alta podria anellar de forma no especifica a temperatures baixes mentre que el oligonucleòtid amb la Tm més baixa pot ser que no anelle de manera eficient a temperatures més altes.
Les temperatures de fusió dels oligonucleòtids es calculen d’una manera molt exacta amb càlculs termodinàmics usant la següent formula:
Tm oligonucleòtids = ΔH [ΔS+ R ln (c/4)] – 273.15°C + 16.6 log 10 [K+]On H és l’entalpia i S l’entropia per a la formació de l’hèlice, R és la constant molar i c és la concentració de l’oligonucleòtid. No obstant això, qualsevol dels programes per al disseny de oligonucleòtids del mercat realitzen est calcule d’una manera ràpida i senzilla.
En qualsevol cas, una bona aproximació (generalment vàlida per a oligonucleòtids en el rang de 18 – 24 bases) es pot calcular amb una aproximació denominada fórmula de Wallace:
Tm oligonucleòtids = 2(A+T) + 4(G+C)Crea una taula on es mostren valors de Tm d’oligonucleòtids de diverses grandàries usant la fórmula de Wallace assumint un contingut en G/C del 50%.
Crea una taula on es mostren valors de Tm de oligonucleòtids de diverses grandàries usant la formula de Wallace assumint un contingut en G/C del 50%.
| Tamany (bases) | Tm = 2(A+T) + 4(G+C) |
|---|---|
| 4 | 12ºC |
| 8 | |
| 10 | |
| 12 | |
| 14 | |
| 16 | |
| 18 | |
| 20 | |
| 22 | |
| 24 | |
| 26 |
Per tant, com es pot observar, la temperatura de fusió d’un oligonucleòtid depèn principalment de la seua longitud i del contingut en G+C. Així, la composició de bases dels oligonucleòtids hauria de ser del 45% al 55% en G/C. A més, la seqüència de l’oligonucleòtid hauria de triar-se de manera que no continga zones de poli G o poli C que poden conduir a hibridació no especifica. De la mateixa manera, cal evitar també les zones riques en poli A i poli T ja que aquestes regions poden provocar una prematura separació dels brins durant el procés d’anellament, la qual cosa conduiria al trencament del complex motle/oligonucleòtid, reduint l’eficiència d’amplificació.
Grandària del oligonucleòtid
La longitud de la seqüència d’un oligonucleòtid influeix en l’especificitat, en la temperatura de fusió i en el temps necessari per a la hibridació del oligonucleòtid a la seua seqüència complementària, per tant és decisiu perquè isca bé la PCR.
La grandària de l’oligonucleòtid és proporcional a l’eficiència d’hibridació: en general, com més llarg siga l’ oligonucleòtid més ineficient serà la hibridació. Si hi ha pocs motles amb el seu oligonucleòtid hibridat (o anellat) en cada pas de la PCR es generaren pocs productes amplificats.
L’associació de cadenes d’àcids nucleics majors de 16pb és un procés extremadament específic. Si tenim en compte les possibilitats combinatòries de diverses “seqüències sonda” donada una cadena de DNA:
| Sonda | Probabilitat | | A, C , G o T/U | 1/4 | | Dinucleótid (ej. AG) | 1/16 | | Tetranucleótid (ej. AGCT) | 1/256 |
D’aquesta manera la probabilitat de trobar una sonda de 20pb és de 1/1.099.511.627.776.
Així doncs, els oligonucleòtids solen tindre un longitud de 18 a 24 bases. Aquesta grandària fa que els oligonucleòtids siguen molt específics de seqüència, sempre que la temperatura d’hibridació siga optima.
Especificitat
L’especificitat de l’oligonucleòtid depèn parcialment de la grandària de l’oligonucleòtid. És evident que un oligonucleòtid de 24 bases és més específic que un de 10 bases. Amb tot, hem de triar oligonucleòtids que tinguen una seqüència única en el motle de DNA que volem amplificar. Per exemple: una parella d’ oligonucleòtids dissenyats amb una seqüència altament repetitiva donarà lloc a multitud de productes de PCR de diferents grandàries quan s’utilitze com a ADN motle l’ADN genòmic d’una espècie. No obstant això, aquests mateixos oligonucleòtid poden donar lloc a una sola banda si amplifiquem un clon d’una genoteca d’ADN.
Atès que la Taq Polimerasa que s’usa en la PCR és activa en un ampli rang de temperatures, simplement que existisca una xicoteta homologia en l’extrem 3’ de l’oligonucleòtid, pot ocórrer una hibridació no especifica del mateix i que tinga lloc una extensió de l’oligonucleòtid a temperatures baixes d’hibridació.
Complementarietat en la seqüència dels oligonucleótid
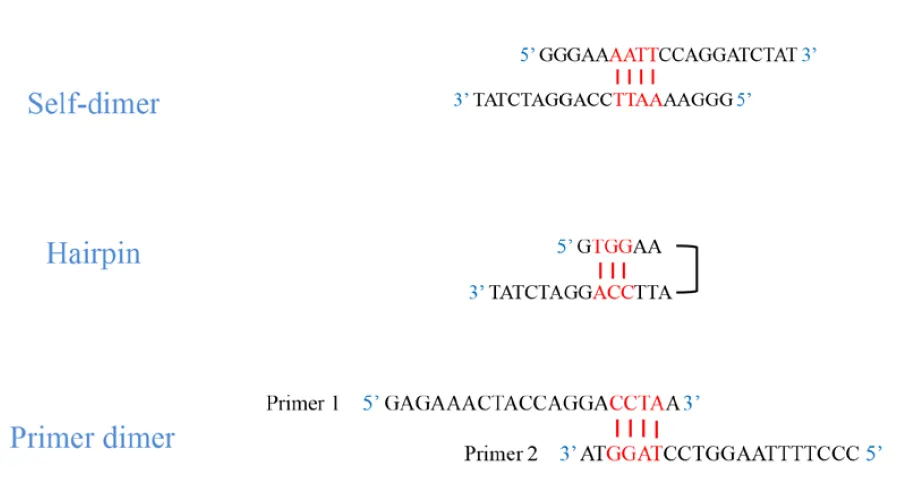
És molt important que els oligonucleòtids no tinguen homologia intra-oligonucleòtid en més de 3 parells de bases. Si un oligonucleòtid té una regió d’auto-homologia, es poden formar estructures parcialment de doble cadena que interferiran amb la hibridació del motle. Aquest tipus d’homologia intra-oligonucleòtid pot donar lloc al fet que es formen estructures secundàries. D’aquesta manera, pot ser que es produïsca tant la unió entre dues cadenes d’un mateix oligonucleòtid (self-dimer), com a la formació d’una forqueta dins de la mateixa cadena de l’oligonucleòtid (hairping).
Un altre perill és l’homologia entre els dos oligonucleòtids. Homologia parcial a les regions mitjanes de dues oligonucleòtids pot interferir amb la hibridació. Si l’homologia ocorreguera en l’extrem 3’ de cada oligonucleòtid, es donarà la formació de dímers de oligonucleòtids (primer-dimer), la qual cosa impedirà que la PCR funcione de manera adequada.
Com a recomanacions, per a cadascuna de les homologies anteriors tindrem en compte:
-
El valor de l’energia lliure de Gibbs (ΔG), que dona una indicació de la força de l’estructura secundària. Es recomana que el valor ΔG sigui més de –9 per a autodimers i heterodimers.
-
Tm o temperatura de fusió, és la temperatura a la qual un dúplex d’oligonucleòtids és del 50% en forma d’una sola cadena i del 50% en forma de doble cadena. Per als hairpins, hauria de ser més baix que la temperatura a la qual s’utilitzarà l’oligo, i sempre la mes baixa possible.
-
Un altre punt a considerar és on es produeix la forqueta, entre els extrems o a les zones del mig. A més si són diversos els punts d’unió o pocs. El millor escenari és que es produïsquen entre les zones del mig i amb pocs punts d’unió.
Temperatura d’anellament (Ta)
La temperatura d’anellament (Ta) és l’estimació de l’estabilitat híbrida d’ADN i ADN híbrid.
Com es va esmentar anteriorment la temperatura de fusió d’una cadena doble d’àcids nucleics està relacionada amb la seua longitud i contingut de G+C. D’aquesta manera la temperatura d’anellament que es tria per a una PCR depén directament de la longitud i composició dels primers. És comú utilitzar temperatures d’anellament 5º per davall de la temperatura de fusió més baixa del parell de primers utilitzats (Innis and Gelfand, 1990).
És important tindre en compte que una Ta massa baixa pot portar a esdeveniments d’inespecificitat dels primers utilitzats, de tal manera que arriben a ser anellats en seqüències diferents a la que volem amplificar . Una Ta massa alta, per part seua, pot portar al fet que el rendiment de l’amplificació siga molt baix ja que es redueix la probabilitat d’anellament.
Fórmula per calcular Ta: Ta = 0,3 x Tm(primer) + 0,7 Tm (producte) – 14,9 on, Tm(primer) = Temperatura de fusió dels primers i Tm(producte) = Temperatura de fusió del producte.
En general, es sol utilitzar una temperatura d’annealització (Ta) de 10º a 15ºC més baixa que la Tm.
Part pràctica
Existeixen una gran varietat de programes, molts d’ells són gratuïts i de fàcil accés a través d’una interfície web. Alguna d’aquestes eines són:
- Oligo (Necessari registre!!)
- Oligo Calc
- Primer designing tool – NCBI
- Netprimer
- Primer3
- eprimer3
- mfeprimer
Durant el desenvolupament de la primera part d’aquesta pràctica, utilitzarem l’eina Primer3 per a dissenyar oligonucleòtids in silico. Primer3 és una aplicació que es troba per al seu lliure ús en diferents servidors web en el món. Utilitzarem la implementació d’aquest programa oferida per University of Tartu & Estonian Biocentre.
La finestra del navegador de Primer3 compta amb diverses seccions que permeten controlar una àmplia varietat de paràmetres (més de 80), relacionats amb el disseny de oligonucleòtids (la majoria d’aqueixos paràmetres rares vegades són modificats).
Pega aquesta seqüència en el camp de text en la part superior de la pàgina de Primer3. Mostra i explica els resultats.
GTTCCGGAATTCGCGCGCGTCGACCCACGCGTCCGTCCGCCTCCACCTACCAACCACCATGGCCACGATCGCTATCATCTACTACTCCACCTACGGCCACATCGCCCAACTGGCCGAGGCCGCCAAGAAGGGTGTCGAATCCGTCGACGGCGTGACCGCCGAGATCTACCAAGTGCAGGAGACTCTCTCCGACGAGATTCTGGGCAAGATGCACGCCGCCCCCAAGAAGGACCACCCCATCGCCACTCTTGACACCCTGAAGGAGGCCGATGGCATCCTGTTCGGTTTCCCCACTCGTTTCGGCTCGCTGCCAGCGCAGGTTAAGGCGTTCTTTGACTCGGCCGGTGGCCTGTGGGCTGCTGGGGCTCTGGTAGGCAAGCCTGCCGGTATCTTCTTCAGCACGGGCACTCAGGGCGGCGGTCAGGAGACAACGGCCTTCACTGCCTTGACGTTCCTAGCTCACCAGGGTCTCACCTTCGTGCCACTGGGCTACCGTGCCCCTGAGCTTTTCAACATGGACGAGCTTCACGGTGGATCCCCGTGGGGAGCGGGAACTCTGGCCGGCGGCGATGGCTCTCGCCAGCCATCGAAGCTGGAGCTGACGGTCGCTACCACCCAGGGTAAGTCGTTCGCTAAAGTGGCCAAGAAGCTGGAGGCTTAAGCACTGCGATTATTTGTGTGTGTGTGTGTCTTGGCTCAAGCTAAATTCTAATGCAATAAAATTTAGTTTTACACGGAra farem servir el Primer designing tool – NCBI per la creació dels primers i Oligo i mfeprimer per a revisar la seua qualitat. Accedeix la seqüència nucleótida corresponent al gen de la insulina humana de Genbank.
-
Quin és el número d’accés de la seqüència?
-
Selecciona els primers que permeten realitzar la PCR per a aquesta seqüència amb l’eina pròpia que et subministra el NCBI, amb els paràmetres que té per defecte. Quants primers hi ha? Sobre que bri d’ADN hibrida forward primer? I el reverse primer?
-
Selecciona, de la taula de resultats el que correspongui amb el darrer digit del teu DNI. Per al primer que has obtingut fes una anàlisi dels següents paràmetres:
-
Quina longitud té el producte d’expressió? Com s’obté el número Product length?
-
Escriu la seqüència del bri codificant que es correspon amb els dos primers. Escriu la seqüència del bri al costat del seu primer, en font de lletra “monospace”. Localitza en la pàgina “Homo sapiens insulin (INS) gene, complete cds”, en l’apartat “Find in this Sequence ” els dos fragments de les seqüències que has trobat. TIP: http://www.bioinformatics.org/sms2/rev_comp.html
-
Quantes guanines i citosines hi ha en cadascun dels primers? I quin és el seu percentatge respecte la seqüència del primer? TIP: http://www.bioinformatics.org/sms2/pcr_primer_stats.html
-
Calcula la temperatura de fusió mitjana dels dos primers utilitzant la fórmula de Wallace.
-
Hi ha homologies internes en les seqüències dels dos primers? Analitza els resultats segons les dades que mostrin l’anal·lisi de l’eina Oligo.
-
-
Descàrrega la seqüència nucleòtida corresponent al gen de la insulina humana de Genbank i selecciona els primers que permeten realitzar la PCR per a aquesta seqüència amb l’eina Primer3. Compara els resultats amb els obtinguts amb l’eina del NCBI.
Dissenya un parell de primers per a la seqüència d’Aedes aegypti rDNA gene Accession “M95126” al NCBI GenBank, de manera que la seqüència de codificació Interna per a la regió trànscrita del Spacer 2 (ITS2) d’aquest gen de l’ADNr s’ampliqui utilitzant una PCR.
Intenta reproduir els estudis del paper “Diseño y evaluación de primers in silico del gen E1 del virus de chikungunya para Real-Time PCR (qPCR)” amb les eines que consideres adequades.
Troba un parell de primers que permetin amplificar per PCR l’exó 10 del gen CFTR en humans. En quines enfermetats es troba involucrat aquest gen? Consells: Utilitzar eines Primer designing tool – NCBI, Oligo i mfeprimer.
EXERCICI PRIMERS PCR
Enunciat i passos a seguir
Trobar primers de PCR (petites seqüències d’ADN complementàries per amplificar un fragment del gen APOE).
Aquí tens el procés:
- Definir la seqüència del gen APOE (o un fragment).
- Trobar primers candidats: Buscarem parells de seqüències que compleixin criteris típics de disseny de PCR:
- Longitud entre 20 nucleòtids.
- Contingut en GC entre 40-60%. -Temperatura de fusió (Tm) equilibrada pels dos primers. El rang ideal de Tm en aplicacions com la PCR sol estar entre 55-65°C.
- Absència d’estructures secundàries (p. ex., dímers o loops).
- Retornar els millors primers per a la PCR.
Obtenir gen APOE
Gen APOE: https://www.ncbi.nlm.nih.gov/gene/348 des del navegador genòmic podeu descarregar el fitxer FASTA amb la seqüència DNA: NM_000041.4
També es pot buscar el nucleòtid directament: https://www.ncbi.nlm.nih.gov/nuccore/NM_000041.4
Trobar Primers
- Fer funció per calcular el percentatge de GC
- Fer funció per calcular la Temperatura dels Primers (fòrmula de Wallace)
- Fer funció que retorni una llista de Forward Primers de 20 nucleòtids amb GC entre 40-60% i Tª 55-65ºC.
# Funció per calcular el percentatge de GCdef gc_content(seq): """Calcula el percentatge de GC en una seqüència d'ADN.""" g = seq.count('G') c = seq.count('C') return (g + c) / len(seq) * 100
# Funció per calcular la temperatura de fusió (Tm)def melting_temp(seq): """Calcula la Tm segons la fórmula de Wallace (aproximada).""" a = seq.count('A') t = seq.count('T') g = seq.count('G') c = seq.count('C') return 2 * (a + t) + 4 * (g + c)
# Funció per trobar primers candidatsdef find_primers(dna_seq, min_len=18, max_len=25): """Troba parells de primers amb bon %GC i Tm equilibrada.""" candidates = []
for start in range(len(dna_seq) - min_len + 1): for length in range(min_len, max_len + 1): if start + length > len(dna_seq): break primer = dna_seq[start:start + length] gc = gc_content(primer) tm = melting_temp(primer)
if 40 <= gc <= 60: # Percentatge GC ideal candidates.append((primer, gc, tm))
return candidates
# Fragment del gen APOE (exemple)apoe_fragment = "ATGCCGTTGACCTGCTAGCTGACTGGCCGATCGGTA"
# Trobar primers candidatsprimers = find_primers(apoe_fragment)
# Seleccionem els millors dos primers (amb Tm equilibrada)if len(primers) >= 2: forward_primer = primers[0] # Primer candidat reverse_primer = primers[-1] # Últim candidat amb característiques adequadeselse: forward_primer = reverse_primer = None
print(primers)print(forward_primer, reverse_primer)Afegir validacions dimers i hairpins
Dímers
Un dímer es produeix quan un primer té regions complementàries internes que poden unir-se, pot ser entre la seqüència i els primers o entre els 2 primers. El més comú és el dímer 3’, on els últims nucleòtids es poden aparellar amb si mateixos.
Validarem que:
- No hi hagi més de 4 parells consecutius en el 3’.
- Evitem seqüències auto-complementàries.
Hairpins (forquetes)
Un hairpin es forma quan un primer es plega sobre si mateix per complementar-se. El criteri per detectar-los serà:
- Buscarem regions complementàries separades per almenys 3 bases (loop central).
- Evitarem seqüències amb més de 4 parells consecutius.
# Funció per comprovar si dues bases són complementàries (A-T, C-G)def is_complementary(base1, base2): return (base1 == 'A' and base2 == 'T') or (base1 == 'T' and base2 == 'A') or \(base1 == 'C' and base2 == 'G') or (base1 == 'G' and base2 == 'C')
# Funció per detectar dímers (3' dímer)def has_dimer(primer, max_pairs=4): """Comprova si un primer té una regió 3' auto-complementària.""" n = len(primer) for i in range(n - max_pairs): match_count = 0 for j in range(max_pairs): if is_complementary(primer[n - 1 - j], primer[n - 1 - (i + j)]): match_count += 1 else: break if match_count == max_pairs: return True # Té un dímer fort return False
# Funció per detectar hairpins (forquetes)def has_hairpin(primer, min_loop=3, max_stem=4): """Comprova si un primer pot formar una forqueta (hairpin).""" n = len(primer) for i in range(n - min_loop - max_stem): for j in range(max_stem): if i + j + min_loop < n and is_complementary(primer[i], primer[i + j + min_loop]): match_count = 1 for k in range(1, max_stem): if i + k < n and i + j + min_loop + k < n and is_complementary(primer[i + k], primer[i + j + min_loop + k]): match_count += 1 else: break if match_count == max_stem: return True # Té un hairpin fort return False
# Funció millorada per trobar primers de PCR sense dímers ni hairpinsdef find_valid_primers(dna_seq, min_len=18, max_len=25): candidates = []
for start in range(len(dna_seq) - min_len + 1): for length in range(min_len, max_len + 1): if start + length > len(dna_seq): break primer = dna_seq[start:start+length] gc = gc_content(primer) tm = melting_temp(primer)
if 40 <= gc <= 60 and not has_dimer(primer) and not has_hairpin(primer): candidates.append((primer, gc, tm))
return candidates
# Fragment del gen APOE (exemple)apoe_fragment = "ATGCCGTTGACCTGCTAGCTGACTGGCCGATCGGTA"
# Trobar primers candidats sense dímers ni hairpinsvalid_primers = find_valid_primers(apoe_fragment)
# Seleccionem els millors dos primers (amb Tm equilibrada)if len(valid_primers) >= 2: forward_primer = valid_primers[0] # Primer candidat reverse_primer = valid_primers[-1] # Últim candidat amb característiques adequadeselse: forward_primer = reverse_primer = None
forward_primer, reverse_primer