Kekule és una biblioteca de JavaScript de codi obert per a quimioinformàtica centrada en representar, dibuixar, editar, comparar i cercar estructures de molècules
On this page
Dibuixant molecules
Les molécules tenen una estructura tridimensional i es poden representar en 3D.
Ehtanol Panel
Per exemple, a PubChem pots veure l’alcohol etílic ( o etanol) en 3D: Ethanol
També pots veure una representació de la molécula a Molview: Ethanol
Les molècules petites sovint són gràfics planars i es poden dibuixar com una representació bidimensional, amb algunes notacions especials per gestionar la quiralitat.
H H | |H-C-C-O-H | | H HAquesta és una representació molt detallada amb tots aquells hidrògens que s’enganxen als àtoms pesats.
Es pot compactar una mica movent les H al costat de l’àtom pesat:
CH<sub>3</sub>-CH<sub>2</sub>-OH
Els àtoms tenen valències: el carboni té una valència de 4, el que significa que necessita 4 enllaços simples, o 1 enllaç doble i 2 enllaços simples, o 2 enllaços dobles, o 1 enllaç triple i 1 enllaç simple.
Com que escriure tants hidrògens és molt pesat, els químics assumeixen que les valències sempre s’ompliran, en lloc d’enumerar els recomptes d’hidrogen de manera explícita, van decidir utilitzar una representació implícita d’hidrogen, on el nombre d’hidrògens en un àtom és la valència de l’àtom més la càrrega menys la suma dels seus ordres d’enllaç.
A continuació tens un alcohol etílic dibuixat amb hidrògens implícits:
C-C-O
A diferència dels químics generals els químics orgànics també han d’escriure moltes C.
Suposo que ja saps per quin motiu, si no ja pots tornar al principi de l’activitat!
La pràctica estàndard és que els carbonis “normals” no es dibuixen en absolut. Qualsevol final d’enllaç o flexió sense símbol d’element s’assumeix que és un carboni sense càrrega de pes molecular mitjà.
Aquí tens un alcohol etílic tal com ho dibuixaria un químic:
--O
Tot això perquè t’interessa?
-
Si mires l’estructura 2D de l’etanol a PubChem no pensarás que algún error ha d’haver-hi: Etanol
-
Que en química orgànica les molècules estan plenes de H i C.
Kekule
Kekule.js és una biblioteca JavaScript de codi obert per a quimioinformàtica.
Actualment, està centrat en les molècules, centrant-se en proporcionar la capacitat de representar, dibuixar, editar, comparar i cercar estructures de molècules als navegadors web.
Tota la biblioteca està dividida en diversos mòduls, cadascun ofereix diferents característiques i funcions. Els usuaris poden utilitzar la combinació de mòduls per a un propòsit específic.
La taula següent enumera les característiques principals de cada mòdul a Kekule.js que ens interessa:
| Mòdul | Caracterísitques principals |
|---|---|
| Core | Representació de conceptes químics com element, àtom, enllaç, molècula i reacció |
| IO | Llegir/escriure diferents formats de dades químiques. Aixó inclou: Format Kekule JSON/XML, CML, MDL MOL2000/3000, SMILES |
| Render | Proporciona mètodes de renderització de navegador creuat de baix nivell per dibuixar molècules (i altres objectes químics) en el context del navegador web |
Dibuixar mol·lècules i àtoms amb Kekule
Els àtoms i els enllaços són la base de diverses molècules.
Estan representats com objectes a Kekule.js.
Crea un projecte kekule tal com s’explica a “/ts/module/” :
$ mkdir kekule && cd kekuleAfegeix la llibreria kekule:
$ npm add kekuleCrea un fitxer index.html:
<!doctype html><html>
<head> <title>Kekule</title> <meta name="viewport" content="width=device-width, initial-scale=1"> <link rel="stylesheet" href=" https://cdn.jsdelivr.net/npm/bootstrap@5.3.1/dist/css/bootstrap.min.css"> <script type="module" src="index.js"></script></head>
<body> <div id="canvas"/></body>
</html>Arrenca un servidor http:
$ npm install http-server --save-dev$ node_modules/http-server/bin/http-server -p 8000 -c1...A continuació creem el fitxer index.js:
import { Kekule } from './node_modules/kekule/dist/kekule.esm.mjs'
const mol = new Kekule.Molecule();const a1 = (new Kekule.Atom()).setSymbol('C')mol.appendNode(a1);
draw(mol)
function draw(mol) {
const drawBoxWidth = 500 const drawBoxHeight = 500
const condensed = true const canvas = document.getElementById('canvas') var bridge = new Kekule.Render.CanvasRendererBridge()
var context = bridge.createContext(canvas, drawBoxWidth, drawBoxHeight)
var baseCoord = { 'x': drawBoxWidth / 2, 'y': drawBoxHeight / 2 } var options = { moleculeDisplayType: condensed ? Kekule.Render.MoleculeDisplayType.CONDENSED : Kekule.Render.MoleculeDisplayType.SKELETAL, retainAspect: true, autoScale: true, zoom: 4 }
bridge.clearContext(context)
var painter = new Kekule.Render.ChemObjPainter(Kekule.CoordMode.COORD2D, mol, bridge) painter.draw(context, baseCoord, options)}Aquest fitxer té una funció draw que s’ocupa de renderitzar la mólecula.
Obre un navegador a http://localhost:8000/:
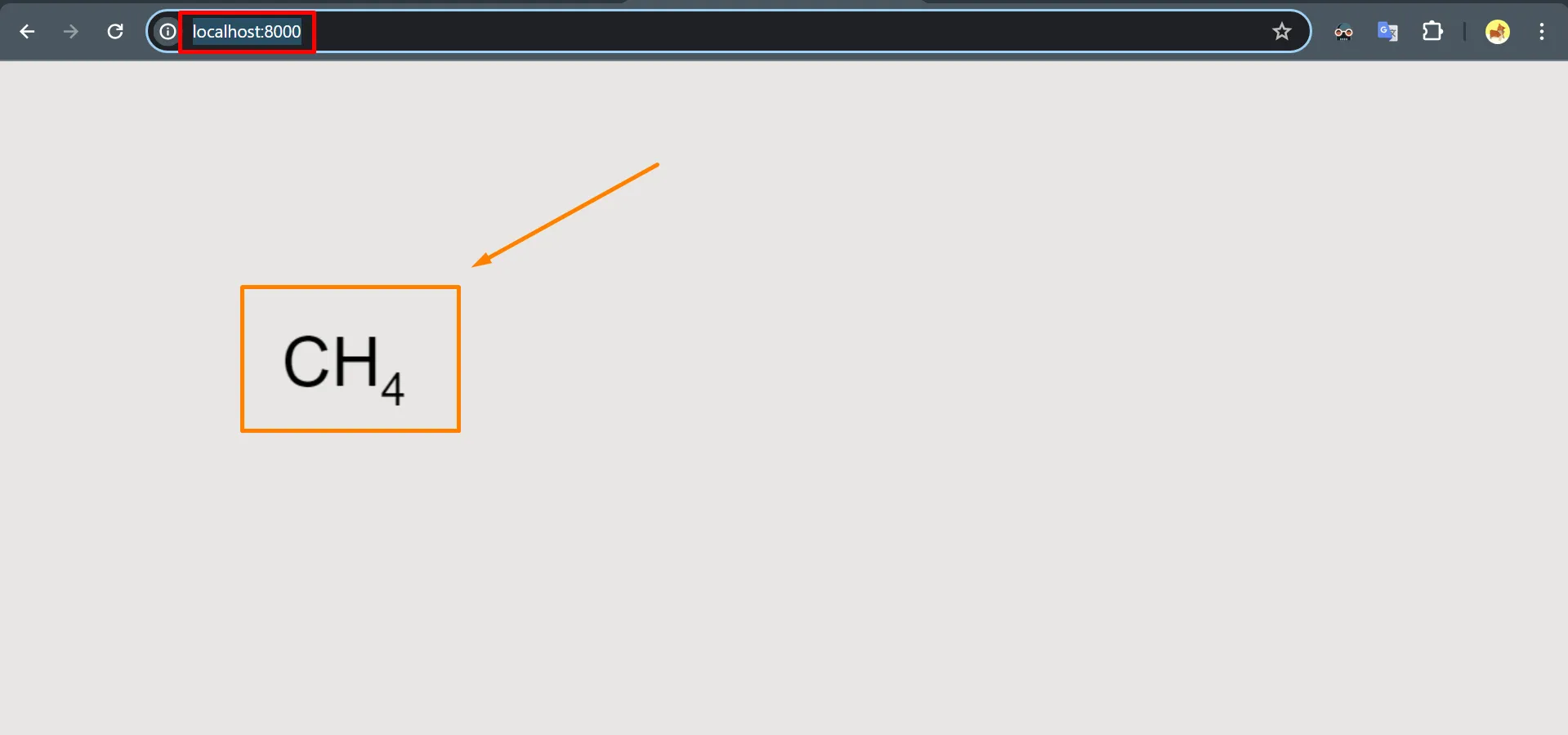
El codi que crea la molécula és aquest:
const mol = new Kekule.Molecule();const a1 = (new Kekule.Atom()).setSymbol('C')mol.appendNode(a1);Llavors com és que apareixen 4 H (hidrogen) ? Suposo que ja saps la resposta (s’ha explicat abans).
Pots provar amb un O (Oxigen) a veure que passa (i pensa perqué passa)
const mol = new Kekule.Molecule();const a1 = (new Kekule.Atom()).setSymbol('O')mol.appendNode(a1);També amb una He (Heli) a veure que passa (i pensa perqué passa).
Pots modificar les propietats de l’àtom:
| Property | ValueType | Descripció |
|---|---|---|
| atomicNumber | int | Nombre atòmic de l’àtom, p. ex. 6 per al carboni. Es canviarà automàticament quan la propietat symbolestà establert. |
| symbol | string | Símbol de l’àtom, per exemple, “C” per carboni. Es canviarà automàticament quan la propietat atomicNumberestà establert. |
| massNumber | int | Nombre de massa de l’isòtop. |
| isotopeId | string | Una propietat de drecera per establir el nombre atòmic i la massa nombre tots alhora, per exemple, ‘13C’ o ‘C13’. |
| charge | float | Càrrega en aquest àtom. Es permet la càrrega parcial (per exemple, +0,5). |
const mol = new Kekule.Molecule();const a1 = (new Kekule.Atom()).setSymbol('C')// set atom to represent C13a1.setSymbol('C').setMassNumber(13); // note that the property can be set cascadedly// same effect as the previous linea1.setIsotopeId('C13');// atom O2-a1.setAtomicNumber(8).setCharge(-2);mol.appendNode(a1);Alguns valors de propietats es poden establir durant la creació de l’àtom:
var atom = new Kekule.Atom('atom1', 'C', 13); // create C13 atomEnllaç
Per crear un vincle:
var bond = new Kekule.Bond('bond1'); // create a bond with an idDues de les propietats més importants de l’enllaç són el seu tipus i ordre:
| Property | ValueType | Descripció |
|---|---|---|
| bondType | string | Tipus de vincle, es pot configurar a: |
Kekule.BondType.COVALENT/IONIC/COORDINATE/METALLIC/HYDROGEN | ||
| bondOrder | int | L’ordre de l’enllaç de covalència es pot establir a: |
kekule.BondOrder.SINGLE/DOUBLE/TRIPLE/… |
// create a double covalent bondbond.setBondType(Kekule.BondType.COVALENT).setBondOrder(2);Si la propietat bondType o bondOrder no està fixada, per defecte es considera que l’enllaç és simple covalent.
Els àtoms es poden connectar entre si mitjançant enllaços, així es crea una molècula.
El codi següent crea una nova molècula d’oxirane amb tres àtoms i tres enllaços:
// create molecule firstconst mol = new Kekule.Molecule()// add three atoms to molecule, property setter can be called cascadelyconst a1 = (new Kekule.Atom()).setSymbol('C').setCoord2D({ 'x': -0.4, 'y': 0.23 })const a2 = (new Kekule.Atom()).setSymbol('C').setCoord2D({ 'x': 0.4, 'y': 0.23 })const a3 = (new Kekule.Atom()).setSymbol('O').setCoord2D({ 'x': 0, 'y': -0.46 })mol.appendNode(a1);mol.appendNode(a2);mol.appendNode(a3);// add three bonds to moleculeconst b1 = (new Kekule.Bond()).setBondOrder(1).setConnectedObjs([a1, a2])const b2 = (new Kekule.Bond()).setBondOrder(1).setConnectedObjs([a2, a3])const b3 = (new Kekule.Bond()).setBondOrder(1).setConnectedObjs([a3, a1])mol.appendConnector(b1)mol.appendConnector(b2)mol.appendConnector(b3)Els àtoms o enllaços també es poden eliminar fàcilment de la molècula existent:
// remove atom O related bonds in moleculemol.removeNodeAt(2); // the atom index starts from 0 // or mol.removeNode(mol.getNodeAt(2));mol.removeConnectorAt(1); // or mol.removeConnector(mol.getConnectorAt(1));mol.removeConnectorAt(2);Molècula després d’eliminar l’àtom O:
[TODO] dibuix
A part dels àtoms normals, també s’admeten pseudoàtoms i àtoms variables:
const mol = new Kekule.Molecule();
// add atoms to moleculemol.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: 0, y: 0.80}));// explicit set mass number of an atommol.appendNode(new Kekule.Atom().setSymbol('C').setMassNumber(13).setCoord2D({x: -0.69, y: 0.40}));mol.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: -0.69, y: -0.40}));// a pseudo atommol.appendNode(new Kekule.Pseudoatom().setAtomType(Kekule.PseudoatomType.ANY).setCoord2D({x: 0, y: -0.80}));mol.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: 0.69, y: -0.40}));mol.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: 0.69, y: 0.40}));// a variable atommol.appendNode(new Kekule.VariableAtom().setAllowedIsotopeIds(['F', 'Cl', 'Br']).setCoord2D({x: 1.39, y: 0.80}));
// add bonds to molecule// here a shortcut method appendBond(atomIndexes, bondOrder) is usedmol.appendBond([0, 1], 1);mol.appendBond([1, 2], 2);mol.appendBond([2, 3], 1);mol.appendBond([3, 4], 2);mol.appendBond([4, 5], 1);mol.appendBond([5, 0], 2);mol.appendBond([5, 6], 1);Fins i tot el subgrup:
const mol = new Kekule.Molecule()
// add atoms to moleculemol.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: 0, y: 0.80}));mol.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: -0.69, y: 0.40}));mol.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: -0.69, y: -0.40}));mol.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: 0, y: -0.80}));mol.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: 0.69, y: -0.40}));mol.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: 0.69, y: 0.40}));
// create a sub groupconst sub = new Kekule.SubGroup();// add atoms/bonds to sub structuresub.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: 1.39, y: 0.80}));sub.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: 2.0, y: 0.40}));sub.appendBond([0, 1], 1);// atom 0 in subgroup connected with main bodysub.appendAnchorNode(sub.getNodeAt(0));sub.setAbbr('Et');
// then add sub structure to moleculemol.appendNode(sub);
// add bonds to molecule// here a shortcut method appendBond(atomIndexes, bondOrder) is usedmol.appendBond([0, 1], 1);mol.appendBond([1, 2], 2);mol.appendBond([2, 3], 1);mol.appendBond([3, 4], 2);mol.appendBond([4, 5], 1);mol.appendBond([5, 0], 2);mol.appendBond([5, 6], 1); // bond connecting subgroupEnllaços multicèntrics
El conjunt d’eines admet enllaços multicèntrics (per exemple, enllaç B-H-B en diborà i enllaç Cp-Fe en ferrocè).
Aquest enllaç es pot crear assignant múltiples àtoms connectats:
// create moleculeconst mol = new Kekule.Molecule();
// add atoms to moleculemol.appendNode(new Kekule.Atom().setSymbol('B').setExplicitHydrogenCount(2).setCoord2D({x: -1, y: 0}));mol.appendNode(new Kekule.Atom().setSymbol('B').setExplicitHydrogenCount(2).setCoord2D({x: 1, y: 0}));mol.appendNode(new Kekule.Atom().setSymbol('H').setCoord2D({x: 0, y: 1}));mol.appendNode(new Kekule.Atom().setSymbol('H').setCoord2D({x: 0, y: -1}));// add two multicenter bond: B-H-Bmol.appendBond([0, 2, 1], 1);mol.appendBond([0, 3, 1], 1);Connexió enllaç-enllaç
També s’admet una connexió especial d’enllaç (per exemple, a la sal de Zeise):
// create moleculevar mol = new Kekule.Molecule();
var atomPt = new Kekule.Atom(); // Pt atommol.appendNode(atomPt.setSymbol('Pt').setCoord2D({x: 0.35, y: 0}));mol.appendNode(new Kekule.Atom().setSymbol('Cl').setCoord2D({x: 0.35, y: 0.80}));mol.appendNode(new Kekule.Atom().setSymbol('Cl').setCoord2D({x: 0.35, y: -0.80}));mol.appendNode(new Kekule.Atom().setSymbol('Cl').setCoord2D({x: 1.14, y: 0}));mol.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: -0.45, y: 0.40}));mol.appendNode(new Kekule.Atom().setSymbol('C').setCoord2D({x: -0.45, y: -0.40}));
mol.appendBond([1, 0], 1, Kekule.BondType.IONIC); // Pt-Clmol.appendBond([2, 0], 1, Kekule.BondType.IONIC); // Pt-Clmol.appendBond([3, 0], 1, Kekule.BondType.IONIC); // Pt-Clvar doubleBond = mol.appendBond([4, 5], 2); // C=C// create bond-bond connectionvar coordinateBond = new Kekule.Bond(); // (C=C)-PtcoordinateBond.setBondType(Kekule.BondType.COORDINATE);coordinateBond.setConnectedObjs([doubleBond, atomPt]);mol.appendConnector(coordinateBond);Fitxers
És molt poc habitual crear una molècula mitjançant codis JavaScript purs.
Normalment l’usuari carrega una molècula de dades externes (per exemple, un fitxer o un string)
El mòdul Kekule.IO proporciona una sèrie de mètodes per realitzar l’entrada i sortida de molècules (i altres objectes químics).
Guardar una mòlecula
Per guardar una molècula, només cal utilitzar saveFormatData:
var data = Kekule.IO.saveFormatData(mol, 'mol'); // or Kekule.IO.saveMimeData(mol, 'chemical/x-mdl-molfile');console.log(data);
var data = Kekule.IO.saveFormatData(mol, 'smi'); // or Kekule.IO.saveMimeData(mol, 'chemical/x-daylight-smiles');console.log(data);Les dades de retorn solen ser un string.
Carregant desde string
Si la font de dades és un string el procés de càrrega és bastant senzill amb Kekule.IO.loadFormatData:
var cmlData = `<cml xmlns="http://www.xml-cml.org/schema"><molecule id="m1"> <atomArray> <atom id="a2" elementType="C" x2="7.493264658965051" y2="35.58088907877604"/> <atom id="a3" elementType="O" x2="8.186084981992602" y2="35.18088907877604"/> <atom id="a1" elementType="C" x2="6.800444335937501" y2="35.18088907877604"/> </atomArray> <bondArray> <bond id="b2" order="S" atomRefs2="a2 a3"/> <bond id="b1" order="S" atomRefs2="a2 a1"/> </bondArray></molecule></cml>`;
var mol = Kekule.IO.loadFormatData(cmlData, 'cml'); // or Kekule.IO.loadMimeData(cmlData, 'chemical/x-cml');La molècula llegida de la cadena es retornarà immediatament. Per exemple, la molècula d’etanol carregada a l’exemple anterior:
Carregant des del fitxer
És una sol·licitud habitual per carregar molècules des del fitxer font.
El conjunt d’eines proporciona dos mètodes per a aquesta tasca: Kekule.IO.loadUrlData i Kekule.IO.loadFileData.
loadUrlData
El mètode loadUrlData obté dades d’un URL de fitxer remot, ja sigui una ruta absoluta (com ara http://www.mysite.com/myMolecule.cml ) o un camí relatiu (com data/myMolecule.cml ). El mètode carrega el fitxer font mitjançant AJAX , així que comparteix les mateixes limitacions amb AJAX (per exemple, same-origin policy.
Com que la transferència de dades a través de la xarxa pot costar molt de temps, loadUrlData utilitzar el model asíncron per retornar el resultat.
Accepta dos paràmetres, el primer és l’URL de la font, la segona és una funció de devolució de trucada que es cridarà quan finalitzi el procés de càrrega. L’usuari hauria de comprovar la molècula carregada en aquesta funció de devolució de trucada:
var url = 'data/mol2D/quinone.mol';Kekule.IO.loadUrlData(url, function(mol, success){ if (success) { console.log('Loading from ' + url + ' Successful'); showMolecule(mol); } else { console.log('Loading from ' + url + ' Failed'); }});loadFileData
En els navegadors web moderns, també es pot carregar el fitxer desde una font local amb Kekule.IO.loadFileData i File API.
Aquest és l’exemple:
document.getElementById('inputFile').addEventListener('change', function() { var file = document.getElementById('inputFile').files[0]; if (file) { Kekule.IO.loadFileData(file, function(mol, success) { if (success && mol) showMolecule(mol); }); } });loadFileDatatambé utilitza el model asíncron i necessita una callback function.
Draw
Per dibuixar fem servir una classe especial que s’anomena painter.
El següent fragment de codi s’utilitza per dibuixar molècules amb Painter:
var renderType = Kekule.Render.RendererType.R2D//R3D // do 2D or 3D drawing
// parent element, we will draw inside itvar parentElem = document.getElementById('parent');// clear parent elemKekule.DomUtils.clearChildContent(parentElem);
// create painter, bind with moleculevar painter = new Kekule.Render.ChemObjPainter(renderType, mol);
// create context inside parentElemvar dim = Kekule.HtmlElementUtils.getElemOffsetDimension(parentElem); // get width/height of parent elementvar context = painter.createContext(parentElem, dim.width, dim.height); // create context fulfill parent element
// at last, draw the molecule at the center of contextpainter.draw(context, {'x': dim.width / 2, 'y': dim.height / 2});De fet, les molècules i altres tipus d’objectes es poden dibuixar a la pàgina HTML d’aquesta manera.
Per exemple, el codi següent dibuixa una fletxa d’equilibri:
// Create arrow glyph with initial parametersvar glyph = new Kekule.Glyph.StraightLine('glyph1', 1, { 'startArrowType': Kekule.Glyph.ArrowType.OPEN, 'startArrowSide': Kekule.Glyph.ArrowSide.REVERSED, 'startArrowWidth': 0.25, 'startArrowLength': 0.25, 'endArrowType': Kekule.Glyph.ArrowType.OPEN, 'endArrowSide': Kekule.Glyph.ArrowSide.SINGLE, 'endArrowWidth': 0.25, 'endArrowLength': 0.25, 'lineLength': 1.5, 'lineGap': 0.1, 'lineCount': 2 });// create new painter, bind with glyphvar painter = new Kekule.Render.ChemObjPainter(Kekule.Render.RendererType.R2D, glyph);// draw the glyph at the center of context we previous createdpainter.draw(context, {'x': dim.width / 2, 'y': dim.height / 2});El resultat:
Formats de fitxers
SDF
El format de fitxer de dades d’estructura (SDF) es basa en el format de fitxer MOL, ambdós desenvolupats per MDL Information Systems que després va ser adquirit per Biovia (ara anomenada Biovia, que pertany a Dassault Systems).
El fitxer MOL original només codificava una sola molècula, mentre que els fitxers en format SDF poden codificar una o diverses molècules.
Els fitxers SDF són fitxers ASCII amb format que emmagatzemen informació sobre les posicions dels àtoms individuals que formen la molècula. També es codifica la informació sobre l’estat d’hibridació i la connectivitat, tot i que aquestes últimes dades s’utilitzen amb menys freqüència i sovint de manera inconsistent.
Múltiples molècules estan delimitades per línies que consisteixen en quatre signes de dòlar ($$$$).
En aquest enllaç tens un exemple d’una molécula d’aigua: https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/name/water/SDF
962 -OEChem-06162407452D
3 2 0 0 0 0 0 0 0999 V2000 2.5369 -0.1550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 3.0739 0.1550 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 2.0000 0.1550 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 1 2 1 0 0 0 0 1 3 1 0 0 0 0M END> <PUBCHEM_COMPOUND_CID>962...Descàrrega directa de mol·lècules.
$ curl https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/name/adenine/SDF -o adenine.molEn general, per a mol·lècules que existeixin les podem baixar amb la instrucció:
curl https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/name/{nom_molecula}/SDF -o {nom_molecula}.molPer descarregar-vos molècules des de TypeScript o Javascript ho podeu fer amb les classes que proporciona el mòdul Kekule.IO:
var reader = new Kekule.IO.MdlReader(); //var multiple = parseInt(document.getElementById('editMultiple').value); var mol1; var result; var r = reader.readData(data, Kekule.IO.ChemDataType.TEXT); mol1 = r; result = r;Informació addicional des del web de l’NCBI:
El subportal d’NCBI pubchem et permet cercar tota la informació sobre mol·lècules:
Quan el trobem, podem veure tota la informació, descarregar-la en diversos formats i fins i tot visualitzar-la amb 2D i 3D. A més a més, et pots descarregar la visualització 2D o 3D.
En el cas de l’Ethanol (l’alcohol), aquestes són les URL que tenim (qualsevol de les 2 val).
El paracetamol: https://pubchem.ncbi.nlm.nih.gov/compound/1983
MOL
Molfile és un format de fitxer basat en text utilitzat per emmagatzemar i transmetre estructures moleculars.
Tot i que els molfiles s’utilitzen més habitualment en química orgànica, el format té èxit en diferents graus a l’hora de representar també molècules inorgàniques i organometàl·liques.
Projecte
Per treballar amb la llibreria farem servir “/ts/react/”
$ git clone https://gitlab.com/xtec/bio-kekule$ cd bio-kekule$ ./init.shTanca el terminal i torna’l a obrir:
$ npm run next-devObre el navegador a http://localhost:3000/molecule.
A partir del projecte inicial has de realitzar modificacions per ampliar la seva funcionalitat.
Per exemple:
-
Opció de renderitzar les molècules en 3D
-
A part de mostrar la molècula, també una descripció de la molécula que pots renderitzar a partir de wikipedia.
-
Que l’usuari pugui modificar el fitxer SDF i es dibuixi la nova molècula
-
Cada cop que es renderitza una molècula enviar el nom al servidor per saber quines són les més buscades.
-
Poder editar una molècula
En aquest enllaç tens vàries demos a partir de kekule: Kekule.js Demos
Aquí tens un altre exemple: Hack-a-Mol
I segur que en troves d’altres a Internet de visualització i manipulació de molècules.
Al finalitzar l’activitat has de construir una imatge i desplegar-la a Azure.
Informació de la mol.lecula
TODO
Incorporar: Atoms, Molecules, and Ions