Uniprot és una base de dades de seqüències de proteïnes de lliure accés amb moltes entrades derivades de projectes de seqüenciació de genomes.
On this page
Introducció
UniProt (Universal Protein Resource) és una base de dades de seqüències de proteïnes i la seva corresponent informació funcional. És de lliure accés i conté moltes entrades derivades de projectes de seqüenciació de genomes.
Conté al voltant de 60 milions de seqüències de proteïnes, derivada de la literatura científica, sobre la funció biològica de les proteïnes, la qual s’actualitza a mesura que es genera més coneixement
Uniprot neix del consorci UniProt que està format per EBI (European Bioinformatic Institute), SIB (Swiss Institute of Bioinformatics, que té una base de dades anomenada Swiss-prot), organitzacions bioinformàtiques europees i PIR (Protein Information Resource) organització americana de dades de proteïnes.
UniProt ofereix accés a quatre bases de dades de proteïnes:
- The UniProt Knowledgebase (UniProtKB),
- The UniProt Reference Clusters (UniRef),
- The UniProt Metagenomics
- Environmental Sequences database
Ves a la pàgina web i mira tota la informació que tens disponible: https://www.uniprot.org/
REST API
UniProt proporciona diverses interfícies de programació d’aplicacions (API) per consultar i accedir a les seves dades mitjançant programes..
UniProt website REST API proporciona URLs RESTful que es poden marcar, enllaçar i utilitzar en programes per a totes les entrades, consultes i eines disponibles a través d’aquest lloc web.
Les dades estan disponibles en tots els formats proporcionats al lloc web, per exemple text, XML, RDF, FASTA, GFF o TSV per a dades de proteïnes UniProtKB.
Obtenir un recurs
L’adreça web d’una entrada consta d’un nom de conjunt de dades (per exemple, uniprot, uniref, uniparc, taxonomy,…) i l’identificador únic de l’entrada, per exemple:

https://www.uniprot.org/uniprotkb/P12345
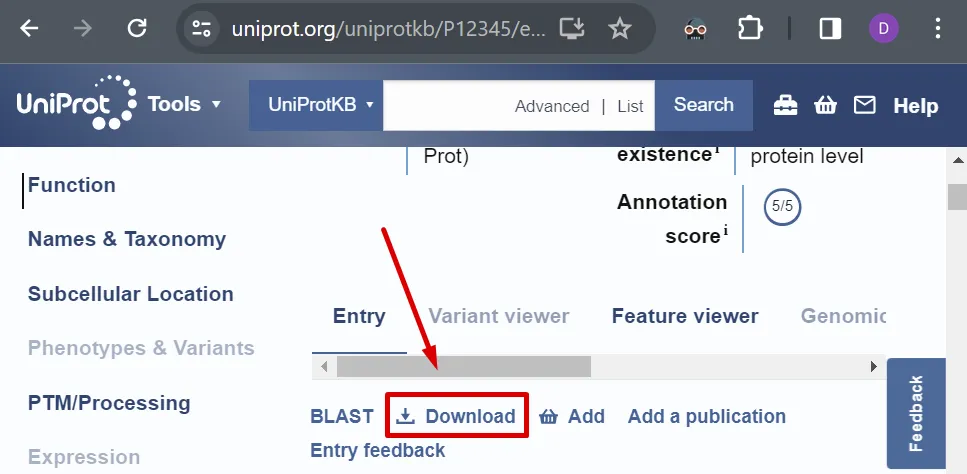
Per defecte, es retorna una pàgina web.
Depenent del conjunt de dades, també poden estar disponibles altres formats (fes clic a “Download” a la pàgina web de l’entrada).
A continuació tens alguns exemples de consultes HTTP amb HTTPX:
import httpximport json
response = httpx.get("https://rest.uniprot.org/uniprotkb/P12345.json")print(json.dumps(response.json(), indent=4))Si coneixes l’estructura del document pots accedir directament a la informació que vols, per exemple la seqüència d’aminoàcids.
import httpximport json
from jsonquerylang import jsonquery
response = httpx.get("https://rest.uniprot.org/uniprotkb/P12345.json")data = response.json()sequence = jsonquery(data, ".sequence")print( json.dumps(sequence, indent=2))Pots veure com és de fàcil accedir a la informació:
{ "value": "MALLHSARVLSGVASAFHPGLAAAASARASSWWAHVEMGPPDPILGVTEAYKRDTNSKKMNLGVGAYRDDNGKPYVLPSVRKAEAQIAAKGLDKEYLPIGGLAEFCRASAELALGENSEVVKSGRFVTVQTISGTGALRIGASFLQRFFKFSRDVFLPKPSWGNHTPIFRDAGMQLQSYRYYDPKTCGFDFTGALEDISKIPEQSVLLLHACAHNPTGVDPRPEQWKEIATVVKKRNLFAFFDMAYQGFASGDGDKDAWAVRHFIEQGINVCLCQSYAKNMGLYGERVGAFTVICKDADEAKRVESQLKILIRPMYSNPPIHGARIASTILTSPDLRKQWLQEVKGMADRIIGMRTQLVSNLKKEGSTHSWQHITDQIGMFCFTGLKPEQVERLTKEFSIYMTKDGRISVAGVTSGNVGYLAHAIHQVTK", "length": 430, "molWeight": 47409, "crc64": "12F54284974D27A5", "md5": "CF84DAC1BDDD05632A89E4C1F186D0D3"}Accedeix a la informació de la seqüència sense utilitzar JSON Query
Consulta la seqüència de l’hemoglobina beta en els humans, que s’encarrega de transportar oxigen a la sang.
https://www.uniprot.org/uniprotkb/P68871
Obtenir un fitxer .fasta
Si només vols la informació de la seqüència pots demanar el resultat en un fitxer FASTA:
from pathlib import Path
import httpx
id = "P99999"
response = httpx.get(f"https://rest.uniprot.org/uniref/UniRef90_{id}.fasta")
path = Path(f"data/{id}.fasta")path.parent.mkdir(parents=True, exist_ok=True)path.write_text(response.text)Aquest cop fem servir el conjunt de dades uniref, enlloc de uniprokb, i un identificador UniRef.
Has de tenir en compte que no es pot garantir que els identificadors UniRef siguin estables, ja que els clusters de seqüències es tornen a calcular a cada “release” i la proteïna representativa pot canviar.
En aquest document t’expliquen com has de crear els “links” a les entrades UniProt: How to link to UniProt entries (UniProtKB, UniParc and UniRef)
Format fasta
El format que veus s’anomena fasta i és dels més comuns en el món bioinformàtic, ja que ens proporciona molta informació de forma compacta i senzilla de tractar.
Qualsevol mena de seqüència o conjunt de seqüències es pot posar en un fitxer FASTA: els 3.000 milions de parells de bases del genoma humà complet, la seqüència d’aminoàcids completa d’un virus o la seqüència d’ADN del promotor del gen del teu ratolí favorit són exemples vàlids.
Si vols descarregar-la per treballar amb ella el més habitual és refer l’URL per tal que et retorni el format textual .fasta
Aquesta URL https://rest.uniprot.org/uniprotkb/P12345.fasta et retorna aquest fitxer:
>sp|P12345|AATM_RABIT Aspartate aminotransferase, mitochondrial OS=Oryctolagus cuniculus OX=9986 GN=GOT2 PE=1 SV=2MALLHSARVLSGVASAFHPGLAAAASARASSWWAHVEMGPPDPILGVTEAYKRDTNSKKMNLGVGAYRDDNGKPYVLPSVRKAEAQIAAKGLDKEYLPIGGLAEFCRASAELALGENSEVVKSGRFVTVQTISGTGALRIGASFLQRFFKFSRDVFLPKPSWGNHTPIFRDAGMQLQSYRYYDPKTCGFDFTGALEDISKIPEQSVLLLHACAHNPTGVDPRPEQWKEIATVVKKRNLFAFFDMAYQGFASGDGDKDAWAVRHFIEQGINVCLCQSYAKNMGLYGERVGAFTVICKDADEAKRVESQLKILIRPMYSNPPIHGARIASTILTSPDLRKQWLQEVKGMADRIIGMRTQLVSNLKKEGSTHSWQHITDQIGMFCFTGLKPEQVERLTKEFSIYMTKDGRISVAGVTSGNVGYLAHAIHQVTKLa primera línia, la que comença per > compte una descripció breu de la cadena.
A partir de la segona línia tenim la seqüència d’aminoàcids, normalment separada per espais \n per facilitar la lectura.
Hi ha fitxers fasta que poden tenir moltes cadenes >, anomenats multifasta. Tractar-los se surt del propòsit de l’activitat.
Tractament amb Python
La web ja informa de quants AA (aminoàcids) tenen les cadenes, però podem obtenir molta més informació amb un programa Python.
Crea i executa aquest codi en un script python, es pot dir fasta.py:
import osimport httpx
uniprot_id = "P12345" # substitueix pel teu codi UniProturl = f"https://rest.uniprot.org/uniprotkb/{uniprot_id}.fasta"fasta_file = f"{uniprot_id}.fasta"
# Descarreguem el fasta en cas de no existir.if not os.path.exists(fasta_file): with httpx.Client(verify=False) as client: response = client.get(url) with open(fasta_file, "w") as f: f.write(response.text) print(f"Descarrega completada: {fasta_file}")
# Obrim el fitxer fasta en mode lectura.with open(fasta_file, "r") as f: fasta_text = f.read()
# Separem les línieslines = fasta_text.splitlines()
# La primera línia és la capçaleraheader = lines[0]
# La resta són la seqüència, la concatenem sense espais ni salts de línia.sequence = "".join(lines[1:])
# Comptar la longitud és molt senzill.longitud = len(sequence)
# Mostrem resultatsprint(f"Capçalera: {header}")print(f"Seqüència: {sequence}")print(f"Longitud: {len(sequence)}")El que fa el programa ho pots llegir als comentaris.
Bàsicament:
- Descarrega el fitxer amb la seqüència en format
.fastaal web d’Uniprot si no existeix - Llegeix el fitxer
.fastaque conté la seqüència. - Separa la capçalera (que està a la primera línia) de la seqüència.
- Mostra la capçalera, la seqüència i la longitud per pantalla.
Obtens el següent resultat:
Capçalera: >sp|P12345|AATM_RABIT Aspartate aminotransferase, mitochondrial OS=Oryctolagus cuniculus OX=9986 GN=GOT2 PE=1 SV=2Seqüència: MALLHSARVLSGVASAFHPGLAAAASARASSWWAHVEMGPPDPILGVTEAYKRDTNSKKMNLGVGAYRDDNGKPYVLPSVRKAEAQIAAKGLDKEYLPIGGLAEFCRASAELALGENSEVVKSGRFVTVQTISGTGALRIGASFLQRFFKFSRDVFLPKPSWGNHTPIFRDAGMQLQSYRYYDPKTCGFDFTGALEDISKIPEQSVLLLHACAHNPTGVDPRPEQWKEIATVVKKRNLFAFFDMAYQGFASGDGDKDAWAVRHFIEQGINVCLCQSYAKNMGLYGERVGAFTVICKDADEAKRVESQLKILIRPMYSNPPIHGARIASTILTSPDLRKQWLQEVKGMADRIIGMRTQLVSNLKKEGSTHSWQHITDQIGMFCFTGLKPEQVERLTKEFSIYMTKDGRISVAGVTSGNVGYLAHAIHQVTKLongitud: 430Exercicis cadenes en Python
Amb els fonaments de programació de Python podem obtenir molta més informació de la cadena.
Per exemple, volem veure quins són els 10 primers caràcters de la seqüència.
Podem fer servir slicing, la mateixa tècnica que feiem servir per seleccionar elements de seqüències (llistes) en Python.
Repassem alguns exemples:

text = "Bon dia!"print(text[0:3]) #Bon!| Expressió | Resultat |
|---|---|
text[0] | 'B' |
text[4:] | 'dia!' |
text[4:7] | 'dia' |
text[1:5] | 'on d' |
text[-1] | '!' |
text[:-1] | 'Bon dia' |
Ara, veiem com aplicar-ho en els nostres fitxers .fasta
Aquest programa llegeix el P12345.fasta (ja descarregat) i mostra els 10 primers caràcters de la seqüència.
fasta_file = "P12345.fasta"
# Obrim el fitxer fasta en mode lectura.with open(fasta_file, "r") as f: fasta_text = f.read()
# Separem les línieslines = fasta_text.splitlines()
# La primera línia és la capçalera (no ens interessa per ara)# header = lines[0]
# Seleccionem únicament la seqüència sense espais ni salts de línia.sequence = "".join(lines[1:])
# Exercici - Mostra els 10 primers caràcters.print(sequence[:10])O volem comptar quantes vegades apareix l’aminoàcid G (la Glicina), seguint el codi anterior.
Per aconseguir-ho usarem el mètode d’utilitat count. Aquí tens un exemple simple:
text = "Hola com va?"print(text.count('a')) #2Ara, just a continuació d’inicialitzar la variable sequence, comptem les G.
# Seleccionem únicament la seqüència sense espais ni salts de línia.sequence = "".join(lines[1:])
# Exercici - Mostra el número de vegades que apareix la G.print(sequence.count('G')) #37 dins del P12345.fastaCompleta l’exemple per tal que mostri: els últims 5 caràcters i els l’intèrval entre el 6 i el 10.
ultims_5 = sequence[-5:]print(ultims_5)del6_al10 = sequence[5:10]print(del6_al10)Completa l’exemple per tal que mostri el número de vegades que apareix la Valina (V). Fes que calculi el % d’aparició de la V respecte el total.
num_v = sequence.count("V")percent_v = (num_v / len(sequence)) * 100print(f"Nombre de Valines (V): {num_v}")print(f"Percentatge de V: {percent_v:.2f}%")Exemple vida real.
Gràcies a aquest domini del llenguatge Python. ja podem identificar mutacions (una alteració de la seqüència d’un organisme que pot tenir efectes negatius en aquest) en cadenes d’aminoàcids.
Exemple: anèmia falciforme
L’hemoglobina (els glòbuls vermells) és la proteïna que transporta oxigen a la sang.
L’anèmia falciforme és una malaltia hereditària en què l’ADN que codifica l’hemoglobina beta (HBB).
Té un únic error en la codificació del sisè aminoàcid de la cadena β de l’hemoglobina (un dels dos tipus de cadenes de proteïnes que conformen l’hemoglobina).
Ho vam veure amb detall a Protein - Structure.
La mutació consisteix en aquest canvi: Glu(E) → Val(V) dins de la posició 6 de la cadena d’hemoglobina.
Per tant, detectar amb Python si una cadena té la mutació o no és senzill:
Primer, baixa la cadena de referència: https://rest.uniprot.org/uniprotkb/P69905.fasta
I executa el codi.
file_fasta = 'P69905.fasta'with open(file_fasta, "r") as f: fasta_text = f.read()
lines = fasta_text.splitlines()header = lines[0]sequence = "".join(lines[1:])
print(f"\nCapçalera: {header}")print(f"Longitud seqüència: {len(sequence)} AA")
# 5️⃣ Mutació anèmia falciforme: Glu -> Val en posició 6 (Hemoglobina)if sequence[5] == "V": print("\nMutació detectada: Glu -> Val a la posició 6")else: print("\nMutació no detectada")Que retorna que, efectivament, la cadena original no està mutada.
Però ja veus que fàcil és comprovar-ho.
Query
Pots utilitzar qualsevol consulta per definir el conjunt d’entrades que t’interessen.
Al principi pot ser més senzill començar amb una cerca de text interactiva al lloc web per trobar l’URL del teu conjunt.
Aquest enllaç et mostra totes les entrades de proteïnes humanes que han estat revisades:
https://rest.uniprot.org/uniprotkb/search?query=(reviewed:true)%20AND%20(organism_id:9606)
Les dades del lloc web les proporciona l’API REST.
Per a l’exemple anterior, la sol·licitud per recuperar el primer lot de dades seria:
response = httpx.get("https://rest.uniprot.org/uniprotkb/search?query=(reviewed:true)%20AND%20(organism_id:9606)")data = response.json()print(json.dumps(data, indent=2))Una consulta no retorna tots els resultats possibles sinó un subconjunt d’aquells més rellevants.
Formats
Per demanar un format concret de dades ho pots fer de diverse maneres.
Paràmetre format
Totes les peticions permeten el paràmetre format, que es pot utilitzar per indicar el format desitjat.
Per exemple, si vull les dades en format tsv:
response = httpx.get("https://rest.uniprot.org/uniprotkb/search?query=reviewed:true+AND+organism_id:9606&format=tsv")data = response.textprint(data)El format TSV és molt útil extreure informació de dades “tabulars”:
Entry Entry Name Reviewed Protein names Gene Names Organism LengthA0A0C5B5G6 MOTSC_HUMAN reviewed Mitochondrial-derived peptide MOTS-c (Mitochondrial open reading frame of the 12S rRNA-c) MT-RNR1 Homo sapiens (Human) 16A0A1B0GTW7 CIROP_HUMAN reviewed Ciliated left-right organizer metallopeptidase (EC 3.4.24.-) (Leishmanolysin-like peptidase 2) CIROP LMLN2 Homo sapiens (Human) 788A0JNW5 BLT3B_HUMAN reviewed Bridge-like lipid transfer protein family member 3B (Syntaxin-6 Habc-interacting protein of 164 kDa) (UHRF1-binding protein 1-like) BLTP3B KIAA0701 SHIP164 UHRF1BP1L Homo sapiens (Human) 1464A0JP26 POTB3_HUMAN reviewed POTE ankyrin domain family member B3 POTEB3 Homo sapiens (Human) 581També pots treballar amb les dades TSV amb Python:
response = httpx.get("https://rest.uniprot.org/uniprotkb/search?query=reviewed:true+AND+organism_id:9606&format=tsv")reader = csv.DictReader(io.StringIO(response.text), dialect="excel-tab")for i, row in enumerate(reader): if i > 10: break print(f"{row['Entry']}\t{row['Entry Name']}")A0A0C5B5G6 MOTSC_HUMANA0A1B0GTW7 CIROP_HUMANA0JNW5 BLT3B_HUMANA0JP26 POTB3_HUMANA0PK11 CLRN2_HUMANA1A4S6 RHG10_HUMANA1A519 F170A_HUMANA1L190 SYCE3_HUMANA1L3X0 ELOV7_HUMANA1X283 SPD2B_HUMANA2A2Y4 FRMD3_HUMANAccept Header
Com és habitual amb les sol·licituds REST, el format desitjat es pot especificar com a capçalera Accept.
Per exemple:
response = httpx.get( "https://rest.uniprot.org/uniprotkb/P12345", headers={"Accept": "text/plain;format=fasta"})print(response.text)Quins formats hi ha disponibles?
Els diferents “end-points” de l’API UniProt proporcionen formats diferents, tot i que, en general, els formats JSON i els valors separats per tabulacions (TSV) estan disponibles en tots els “end-points”.
| Description | Accept | Format |
|---|---|---|
| JavaScript Object Notation (JSON) | application/json | json |
| Extensible Markup Language (XML) | application/xml | xml |
| Text file representation | text/plain;format=flatfile | txt |
| List of one or more IDs | text/plain;format=list | list |
| Tab-Separated-Values | text/plain;format=tsv | tsv |
| FASTA | text/plain;format=fasta | fasta |
| Genomic Feature Format (GFF) | text/plain;format=gff | gff |
| Open Biomedical Ontologies (OBO) | text/plain;format=obo | obo |
| Resource Description Framework (RDF) | application/rdf+xml | rdf |
| Excel | application/vnd.ms-excel | xlsx |
Tips
-
Familiaritza’t amb el advanced search builder fent clic a Advanced.
-
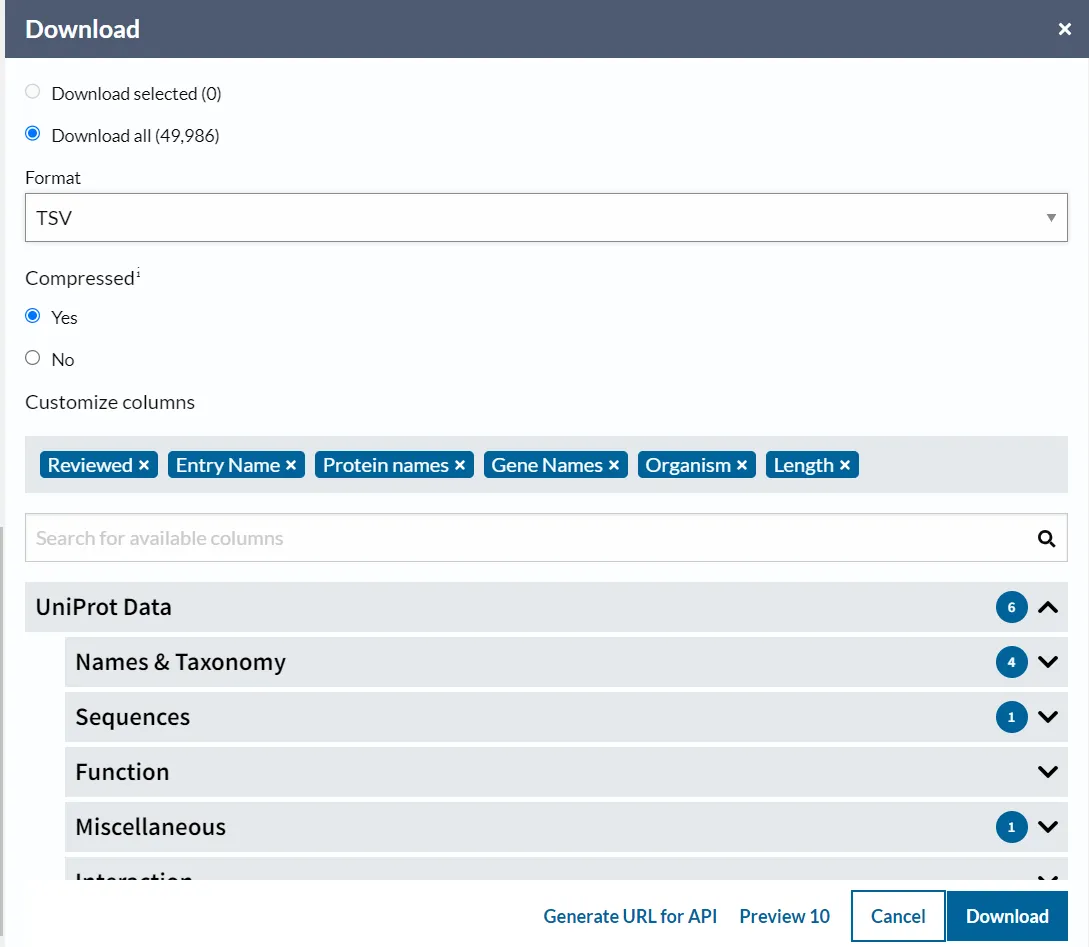
Fes clic a Customize data a la pàgina de resultats de la cerca per seleccionar les columnes que es mostraran a la taula de resultats.
-
També pots cercar els noms de columnes rellevants a la llista completa de UniProtKB column names for programmatic access.
-
Alguns d’aquests formats els pots emmagatzemar en Dataframes.
L’URL del resultat d’una consulta consta d’un nom de conjunt de dades (p. ex., uniprot, uniref, uniparc, taxonomia,…) i la consulta real.
S’admeten els paràmetres de consulta següents:
TODO urls
| Parameter | Values | Description |
|---|---|---|
| query | string | See query syntax and query fields for UniProtKB. |
| An empty query string will retrieve all entries in a data set. Tip: Refine your search by clicking Advanced in the search bar. | ||
| format | See section, “What formats are available?“ | |
| fields | comma-separated list of column names | Columns to retrieve in the results. Applies to tsv, xslx and json formats only. (For UniProtKB you can also read the full list of UniProtKB column names). |
| includeIsoform | true or false | Whether or not to include isoforms in the search results. Note: Only applies to UniProtKB searches. |
| compressed | true or false | Return results gzipped. Note that if the client supports HTTP compression, results may be compressed transparently even if this parameter is not set to true. |
| size | integer | Maximum number of results to retrieve. Note: Only takes effect on searches. |
| cursor | string | Specifies the cursor position in the entire result set, from which returned results will begin. Cursors are used to allow paging through results. Typically used together with the size parameter. |
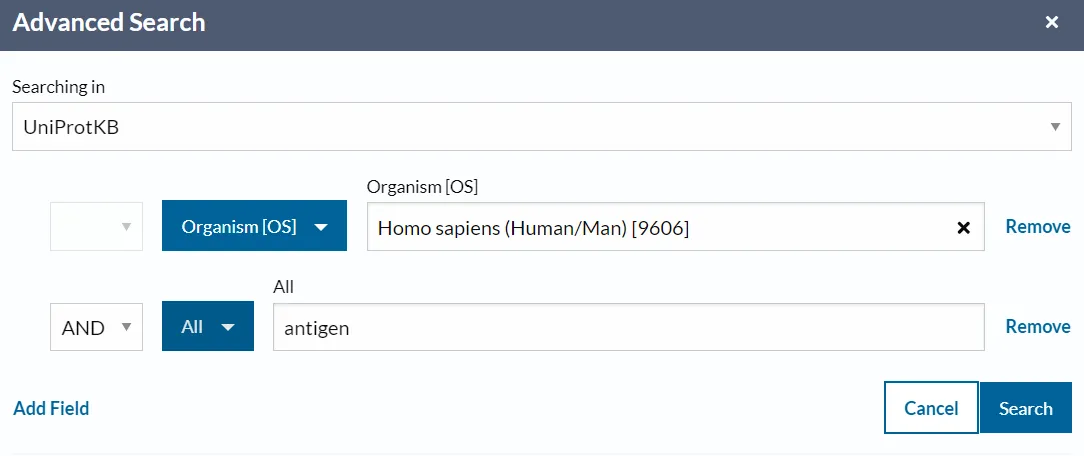
L’exemple següent recupera totes les entrades humanes que coincideixen amb el terme “antigen” en formats TSV comprimit:
$ curl -s "https://rest.uniprot.org/uniprotkb/search?query=reviewed:true+AND+organism_id:9606&format=tsv&compressed=true" > data/organism_9696.tsv.gz...Com que les dades estan comprimides no les pots utilitzar directament. Primer les has de descomprimir:
També pots fer la consulta avançada; però és més lent.
El següent exemple recupera totes les entrades humanes amb referències creuades a PDB en format separat per tabulacions, mostrant només els identificadors UniProtKB i PDB.
$ http -p b "https://rest.uniprot.org/uniprotkb/search?query=organism_id:9606+AND+database:pdb&format=tsv&fields=id,xref_pdb" | head -n 5...El Protein - Protein Data Bank és l’altra gran base de dades de proteïnes que veurem més endavant.
Stream
Si volem que l’API ens torni tots els resultats d’una consulta podem utilitzar l’API stream, encara que només tenim que fer-ho si esperem un nombre “petit” de resultats.
L’objectiu és descarregar les entrades revisades de l’organisme SARS-CoV-2 a UniProtKB.
Farem servir l’”streaming endpoint” que retorna un únic string amb totes les seqüències FASTA.
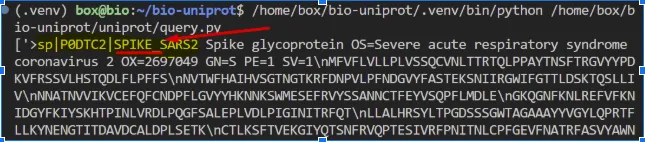
Per trobar l’entrada de l’organisme has de crear una llista de seqüències FASTA i trobar totes les seqüències que a la capçalera fagin menció a SPIKE:
stream-save.py
ìmport reimport urllib.parseimport urllib3
parameters = "format=fasta&query=(organism_id:2697049) AND (reviewed:true)"
#####
parameters = urllib.parse.quote(parameters, safe='=&')url = f"https://rest.uniprot.org/uniprotkb/stream?{parameters}"response = urllib3.request("GET", url)fastas = response.data.decode("utf-8")
# Create a list of FASTA sequencesfastas = re.split(r'\n(?=>)', fastas)# Find all sequences with header mentioning SPIKEfastas = [fasta for fasta in fastas if 'SPIKE' in fasta]
print(fastas)Aquí tens el resultat de la cerca:
Si la vols guardar:
f = open("fastas.fasta", "w")for fasta in fastas: f.write(fasta)f.close()Limitacions
El “stream endpoint” suposa molta càrrega de processament a l’API i per aquest motiu hi ha una limitació en el nombre de sol·licituds paral·leles que gestiona.
En el cas que l’stream API tingui massa sol·licituds, la sol.licitut HTTP pot retorna un estat 429.
Si això passa pots utilitzar la paginació (que veurem a continuació) o tornar a provar més tard.
El “stream endpoint” pot gestionar conjunts de resultats de com a màxim de 10.000.000 d’entrades.
Si necessites un conjunt més gran de dades pots descarregar els fitxers FTP:
query (pagination)
Quan el resultat de la resposta és un gran nombre de resultats, és millor utilitzar la paginació, que vol dir obtenir els resultats d’un en un.
És preferible al streaming perquè:
- Menor ús de la memòria. Si les dades dels resultats de la cerca superen la memòria de l’ordinador, la descàrrega mitjançant streaming farà que el script de Python es bloquegi.
La paginació només carrega un subconjunt dels resultats a la memòria a la vegada.
- L’aplicació és més robusta davant problemes de connexió. Si durant una descàrrega per streaming la connexió s’interromp, la descàrrega s’haurà de reiniciar des del principi.
Quan es treballa amb la paginació, es pot tornar a provar cada lot des del punt de fallada sense necessitat de reiniciar tot el procés.
- Menys demanda de recursos a l’API. El “stream endpoint” requereix una gran quantitat de memòria.
El “pagination endpoint” distribueix la demanda de recursos durant un període de temps més llarg, de manera que la infraestructura de l’API ho pot gestionar millor.
- Pots processar cada lot a mesura que arriba. La descàrrega per streaming requereix que la descàrrega s’hagi completat abans que es pugui processar.
El processament per lots permet intercalar el processament amb la descàrrega, cosa que pot ser útil si es vol veure els resultats processats el més aviat possible.
Aquí tens un diagrama que mostra les diferències:
Stream Pagination+------------+ +------------+| Download 1 | | Download 1 || Download 2 | | Process 1 || Download 3 | | Download 2 || Process 1 | | Process 2 || Process 2 | | Download 3 || Process 3 | | Process 3 |+------------+ +------------+Capçaleres de la resposta
Quan fas una consulta(“query endpoint”) l’API està dissenyada per retornar els resultats en lots (pàgines) amb els resultats més rellevants de la consulta en els primers lots.
Ademés del resultat de les dades del lot, en la capçaleres HTTP de la resposta tens una capçalera link que indica on trobar la següent pàgina de resultats:
curl -sI "https://rest.uniprot.org/uniprotkb/search?query=human%20cdc7" | grep ^link...Resposta:
link: <https://rest.uniprot.org/uniprotkb/search?query=human%20cdc7&cursor=1mkycb2xwxbouwf6pw9zoi9t13q0jdwr1xmg&size=25>; rel="next"També pots veure que la URL es construeix perquè torni 25 resultats per lot (size=25) si en la solicitud HTTP no has indicat un valor específic amb l’argument size.
Aquest procediment és semblant al que es fa a Entrez amb Biopython i el History Server, però molt més senzill ja que la resposta ja inclou montada la propera URL que has d’utilitzar.
Una altra capçalera rellevant és el nombre de resultats de la resposta:
$ curl -sI "https://rest.uniprot.org/uniprotkb/search?query=human%20cdc7" | grep x-total-results...Python
A continuació tenim un programa que busca totes les entrades (articles) revisades que contenen la paraula insulina, ordenades pel nombre més gran d’interaccions.
Pots veure que el programa va fent peticions HTTP (amb una lògica de Retry) fins que arriba una resposta sense la capçalera Link.
També pots veure que com a paràmetre passem el valor size=500 que és el valor màxim admés.
Per obtenir el next link busquem la capçalera Link, i si la trobem, fem servir una expressió regular per extreure la URL:
search.py
import httpx
parametrs = "fields=accession,cc_interaction&format=tsv&query=Insulin AND (reviewed:true)"
#####
client = httpx.Client(follow_redirects=True)
def get_batch(batch_url): while batch_url: response = client.get(batch_url) response.raise_for_status() batch = response.text total = response.headers.get("x-total-results") yield batch, total batch_url = response.links.get("next", {}).get("url")
url = f"https://rest.uniprot.org/uniprotkb/search?{parametrs}&size=500"
interactions = {}for batch, total in get_batch(url): for line in batch.splitlines()[1:]: primaryAccession, interactsWith = line.split('\t') interactions[primaryAccession] = len(interactsWith.split(';')) if interactsWith else 0 print(f'{len(interactions)} / {total}')
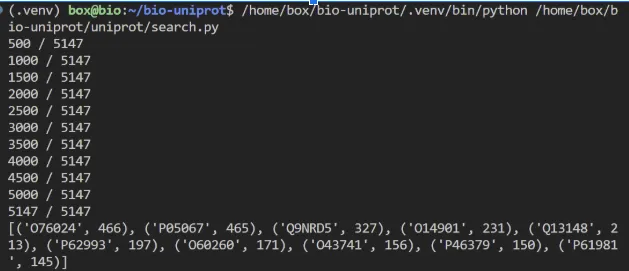
# See the accessions with the greatest number of interactionssorted_interactions = sorted(interactions.items(), key=lambda item: item[1], reverse=True)print(sorted_interactions[:10])A continuació tens el resultat d’executar el programa:
Guardar fitxers
La lògica de guardar fitxers és molt senzilla a partir dels programes anteriors.
stream
urllib3 està preparada per treballar amb streams.
A mida que van arribant bytes d’informació l’anem processat en blocs de 1024 bytes escrivint al fitxer corresponent.
Posteriorment descomprimim el fitxer.
Tot això només ho fem si no tenim el fitxer descarregat i descomprimit.
stream.py
import urllib.parseimport urllib3import gzipimport shutilimport os
input_file = "data/SARS-CoV-2.fasta.gz"output_file = "data/SARS-CoV-2.fasta" # Fitxer descomprimit
if not os.path.exists(input_file): parameters = { "compressed": "true", "format": "fasta", "query": "(organism_id:2697049) AND (reviewed:true)" } encoded_parameters = urllib.parse.urlencode(parameters) url = f"https://rest.uniprot.org/uniprotkb/stream?{encoded_parameters}"
resp = urllib3.request("GET", url, preload_content=False) with open(input_file, 'wb') as file: for chunk in resp.stream(1024): file.write(chunk)
resp.release_conn()
with gzip.open(input_file, 'rb') as f_in: with open(output_file, 'wb') as f_out: shutil.copyfileobj(f_in, f_out)Altres formes d’accés
Unipress
Com que l’API es pot utilitzar amb qualsevol llenguatge de programació és habitual que es vagin creant llibreries per facilitar la interacció amb l’API.
L’última llibreria en Python és aquesta:
nodejs
Com passava amb la base de dades Entrez, també pots interactuar directament amb la Uniprot mitjançant Javascript.
Com que es tracta d’una API REST pots utilitzar una llibreria de client REST.
https://www.npmjs.com/package/node-rest-client
És especialment interessant usar-la si no podem crear-nos una API pròpia (ja que Nodejs fa de servidor, i així prevenim errors de CORS).
Crear webapp per accedir a Uniprot.
Crea la API amb FastAPI
Primer, pots crear una API amb Python i FastAPI.
from fastapi import FastAPI, HTTPExceptionfrom fastapi.responses import JSONResponsefrom pydantic import BaseModelfrom typing import Listimport urllib3import os
app = FastAPI()
@app.get("/")def welcome(): return {"Funciona!"}
## -- ACCÉS A UNIPROT -- ##http = urllib3.PoolManager()
@app.get("/api/protein/")def get_protein_sequence(protein_id: str = None): # Consulta a Uniprot url = f"https://rest.uniprot.org/uniprotkb/{protein_id}.fasta" response = http.request("GET", url) if response.status != 200: raise HTTPException(status_code=404, detail="Proteïna no trobada")
# Convertim la resposta en text i la dividim per seqüències FASTA fasta_data = response.data.decode("utf-8").strip().split("\n>")
# Comprovem si hi ha una sola seqüència sense el ">", i l'afegim si cal if fasta_data[0] and not fasta_data[0].startswith(">"): fasta_data[0] = ">" + fasta_data[0] fasta_data = [">" + seq if not seq.startswith(">") else seq for seq in fasta_data]
return {"protein_id": protein_id, "sequences": fasta_data}Prova-la 🐍 fàcilment a http://0.0.0.0:8000/docs i si funciona passa al següent pas!
La resposta hauria de ser un codi 200 i una resposta com aquesta (que per facilitar la visualització s’han afegit salts de línia \n)
{ "protein_id": "P12345", "sequences": [ ">sp|P12345|AATM_RABIT Aspartate aminotransferase, mitochondrial OS=Oryctolagus cuniculus OX=9986 GN=GOT2 PE=1 SV=2\nMALLHSARVLSGVASAFHPGLAAAASARASSWWAHVEMGPPDPILGVTEAYKRDTNSKKM\nNLGVGAYRDDNGKPYVLPSVRKAEAQIAAKGLDKEYLPIGGLAEFCRASAELALGENSEV\nVKSGRFVTVQTISGTGALRIGASFLQRFFKFSRDVFLPKPSWGNHTPIFRDAGMQLQSYR\nYYDPKTCGFDFTGALEDISKIPEQSVLLLHACAHNPTGVDPRPEQWKEIATVVKKRNLFA\nFFDMAYQGFASGDGDKDAWAVRHFIEQGINVCLCQSYAKNMGLYGERVGAFTVICKDADE\nAKRVESQLKILIRPMYSNPPIHGARIASTILTSPDLRKQWLQEVKGMADRIIGMRTQLVS\nNLKKEGSTHSWQHITDQIGMFCFTGLKPEQVERLTKEFSIYMTKDGRISVAGVTSGNVGY\nLAHAIHQVTK" ]}Crear una web que consulti la API amb React.
Ara, pots crear un client web amb React (o qualsevol tecnologia que usi Javascript).
Aquesta pàgina mostra un formulari on l’usuari li passa el protein_id (per exemple P12345).
import { useState } from "react";
export default function App() { const [proteina, setProteina] = useState<string>(""); const [resultat, setResultat] = useState<string[]>([]); const [error, setError] = useState<string>("");
const consultarProteina = () => { if (!proteina.trim()) { setError("Si us plau, introdueix el codi o nom de la proteïna."); return; } setError("");
// Crida a la API FastAPI del teu backend fetch(`/api/protein/?protein_id=${proteina.trim()}`) .then((resposta) => { if (!resposta.ok) { throw new Error(`Error ${resposta.status}: ${resposta.statusText}`); } return resposta.json(); }) .then((dades) => { setResultat(dades.sequences); }) .catch((err) => { console.error("Error en la consulta:", err); setError("Error en obtenir la informació de la proteïna."); setResultat([]); }); };
return ( <main className="container mt-5"> <h2>Consulta informació de proteïna (UniProt)</h2>
<div className="mb-3"> <label htmlFor="proteina" className="form-label"> Introdueix el codi de la proteïna: </label> <input type="text" className="form-control" id="proteina" value={proteina} onChange={(e) => setProteina(e.target.value)} placeholder="Ex: P12345" /> </div>
<button className="btn btn-primary" onClick={consultarProteina}> Consultar </button>
<div className="resultat mt-4"> {error && <div className="alert alert-danger">{error}</div>} {resultat.length > 0 && ( <> <div className="mt-3"> <label htmlFor="sequencia" className="form-label"> <strong>Seqüències FASTA:</strong> </label> <textarea id="sequencia" className="form-control" value={resultat.join("\n\n")} // Separem per línies buides rows={10} readOnly /> </div>
<a href={`/path/to/fasta/${proteina.trim()}.fasta`} // Aquí afegim la URL per descarregar directament download={`${proteina.trim()}.fasta`} className="btn btn-success mt-3" > Descarregar FASTA </a> </> )} </div> </main> );}Activitats
1.- Fes un programa que descarregui les 5 primeres entrades d’organismes que contenen el gen INS (el de la insulina) des d’Uniprot.
Obtén el fitxer .fasta d’aquestes entrades:
https://www.uniprot.org/uniprotkb?query=gene%3AINSimport urllib3import jsonimport os
# Crear el client de peticions HTTPhttp = urllib3.PoolManager()
# Definir la funció per obtenir les seqüències FASTA dels primers resultatsdef obtenir_gen_ins_fasta():url = "https://rest.uniprot.org/uniprotkb/stream?query=gene%3AINS&format=fasta&size=5"resposta = http.request("GET", url)
if resposta.status != 200:print(f"Error al fer la consulta a UniProt (codi {resposta.status})")return None
return resposta.data.decode("utf-8")
# Funció per desar el fitxer FASTA en una carpeta localdef desar_fasta(fasta_data):if not fasta_data:print("No s'han obtingut dades FASTA.")return
if not os.path.exists("data/fasta_files"):os.mkdir("data/fasta_files")
# Desar les dades FASTA en un fitxerwith open("data/fasta_files/insulin_sequences.fasta", "w") as f:f.write(fasta_data)print("Fitxer 'insulin_sequences.fasta' desat correctament.")
# Obtenir les 5 primeres seqüències FASTA del gen INSfasta_data = obtenir_gen_ins_fasta()desar_fasta(fasta_data)2.- Millora o crea una webapp com la de l’exemple que mostri aquesta informació addicional.
{ "protein_id": "P12345", "name": "Aspartate aminotransferase", "organism": "Oryctolagus cuniculus", "length": 504, "function": "Catalyzes the reversible conversion of aspartate and alpha-ketoglutarate to oxaloacetate and glutamate."}from fastapi import FastAPI, HTTPExceptionimport requestsfrom typing import Listimport os
app = FastAPI()
@app.get("/")def welcome():return {"message": "Benvingut a la API d'Uniprot!"}
## -- ACCÉS A UNIPROT -- ##
@app.get("/api/protein/")def get_protein_data(protein_id: str):url = f"https://rest.uniprot.org/uniprotkb/{protein_id}.json"response = requests.get(url)
if response.status_code != 200:raise HTTPException(status_code=404, detail="Proteïna no trobada")
data = response.json()
protein_info = {"protein_id": protein_id,"name": data.get("protein", {}).get("recommendedName", {}).get("fullName", "No disponible"),"organism": data.get("organism", {}).get("scientificName", "No disponible"),"length": len(data.get("sequence", {}).get("value", "")),"function": data.get("comment", {}).get("function", "No disponible")}return protein_infoimport { useState } from "react";
export default function App() {const [proteinId, setProteinId] = useState<string>("");const [result, setResult] = useState<any>(null);const [error, setError] = useState<string>("");
const consultarProteina = () => {if (!proteinId.trim()) {setError("Si us plau, introdueix un codi de proteïna.");return;}setError("");
// Crida a la API FastAPI del teu backendfetch(`/api/protein/?protein_id=${proteinId.trim()}`).then((response) => {if (!response.ok) {throw new Error(`Error ${response.status}: ${response.statusText}`);}return response.json();}).then((data) => {setResult(data);}).catch((err) => {console.error("Error en la consulta:", err);setError("No s'ha pogut obtenir la informació de la proteïna.");setResult(null);});};
return (<main className="container mt-5"><h2>Consulta informació de proteïna d'Uniprot</h2>
<div className="mb-3"><label htmlFor="proteinId" className="form-label">Introdueix el codi o nom de la proteïna:</label><inputtype="text"className="form-control"id="proteinId"value={proteinId}onChange={(e) => setProteinId(e.target.value)}placeholder="Ex: P12345"/></div>
<button className="btn btn-primary" onClick={consultarProteina}>Consultar Proteïna</button>
<div className="result mt-4">{error && <div className="alert alert-danger">{error}</div>}{result && ( <div className="mt-3"> <h3>Informació de la proteïna {result.protein_id}</h3> <ul> <li><strong>Nom:</strong> {result.name}</li> <li><strong>Organisme:</strong> {result.organism}</li> <li><strong>Longitud:</strong> {result.length} aminoàcids</li> <li><strong>Funció:</strong> {result.function}</li> </ul> </div>)}</div></main>);}