El format Protein Data Bank és un format de fitxer comunament utilitzat per emmagatzemar informació sobre estructures moleculars.
On this page
Introducció
El RCSB Protein Data Bank (RCSB PDB) proporciona accés i eines per a l’exploració, visualització i anàlisi de:
- Estructures 3D determinades experimentalment a partir de l’arxiu del Protein Data Bank (PDB).
- Computed Structure Models (CSM) d’AlphaFold DB i ModelArchive.
- Fins i tot, programes d’anàlisis de proteïnes i predicció d’estructura com FireProt 2.0 i Modeller
Aquesta base de dades és el repositori de referència de dades estructurals de proteïnes, un recurs essencial per a aplicacions com el disseny de fàrmacs i l’enginyeria de proteïnes
Pots accedir als registres d’aquesta base de dades a través de la pàgina web de RCSB a https://www.rcsb.org/
Per exemple, pots visualitzar el registre de l’Hemoglobina humana en 3D, que té el codi 4N7N, en aquest enllaç https://www.rcsb.org/3d-view/4n7n
Els noms dels fitxers PDB consisteixen en un número seguit de tres caràcters, per exemple, 1HEW.
PDB Format
Un fitxer PDB (Protein Data Bank) és un format de fitxer estandarditzat basat en text que emmagatzema les dades estructurals tridimensionals (3D) de macromolècules biològiques, principalment proteïnes i àcids nucleics, així com els seus complexos.
Un fitxer PDB emmagatzema les posicions espacials dels àtoms obtingudes per cristal·lografia de raigs X, espectroscòpia RMN i altres tècniques experimentals, i contenen les coordenades atòmiques, informació d’enllaç i altres detalls rellevants de les biomolècules derivades de mètodes experimentals…
Descarrega el fitxer “Legacy PDB Format” de la proteïna Zeta-Zeta Transmembrane Dimer des del PowerShell de Windows:
iwr https://files.rcsb.org/download/2HAC.pdb -OutFile 2HAC.pdbA Linux seria:
curl -o 2HAC.pdb https://files.rcsb.org/download/2HAC.pdbEl fitxer és un fitxer de text que pots obrir amb l’editor de text.
Title Section
Els fitxers PDB comencen amb un Title Section.
Aquesta secció conté detalls generals sobre les molècules en el fitxer, així com els experiments utilitzats per elucidar les seves estructures. Tot i que hi ha diversos tipus de registres en una secció de títol, els que discutirem a continuació són HEADER, COMPND, i REMARK.
Header Record
La primera línia és un registre header, i té aquest aspecte:
HEADER MEMBRANE PROTEIN 12-JUN-06 2HACEn altres paraules, l’estructura en aquest fitxer és una proteïna de membrana; va ser dipositada el 12 de juny de 2006; i el seu ID PDB és 2HAC.
El més important a notar sobre aquest registre és que la posició de tot dins del registre és important. El format PDB utilitza la posició del caràcter (o “columna”) per delimitar camps dins de cada registre. Això és cert per a tots els tipus de registres, no només per al HEADER. En aquest exemple particular, els primers sis caràcters són el nom del registre ("HEADER"), els caràcters 11-50 estan reservats per al camp de “classificació” (proteïna de membrana), els caràcters 51-59 s’utilitzen per a la data de dipòsit, i els caràcters 63-66 contenen l’ID de l’estructura.
Compound Record
Un dels següents registres en 2hac.pdb és un registre de compost, i té aquest aspecte:
COMPND MOL_ID: 1;COMPND 2 MOLECULE: T-CELL SURFACE GLYCOPROTEIN CD3 ZETA CHAIN;COMPND 3 CHAIN: A, B;COMPND 4 FRAGMENT: TRANSMEMBRANE REGION (28-60);COMPND 5 SYNONYM: T-CELL RECEPTOR T3 ZETA CHAIN;COMPND 6 ENGINEERED: YESEl registre de compost és un exemple d’un únic registre que ocupa múltiples línies. Cada línia consisteix en un parell token/valor, que hauria de recordar-vos el tipus de dades diccionari de Python, o arrays associatius d’altres llenguatges de programació.
Els registres multilínia utilitzen números per permetre la continuació d’un sol registre. Així, per exemple, la línia que comença amb "COMPND 2" simplement continua el registre de compost, donant un altre parell token/valor. En aquest cas, el token (o clau) és MOLECULE i el valor és T-CELL SURFACE GLYCOPROTEIN CD3 ZETA CHAIN.
Remark Record
Poc després del registre de compost hi ha una llista de diversos centenars de línies de remarks. Un comentari PDB és similar als comments en Python o altres llenguatges. En els fitxers PDB, un comentari comença amb REMARK i un número, que simplement identifica el comentari. Per exemple, el comentari #3 comença així:
REMARK 3REMARK 3 REFINEMENT.REMARK 3 PROGRAM : NMRPIPE 1.0, XPLOR-NIH 2.11REMARK 3 AUTHORS : FRANK DELAGLIO (NMRPIPE), CHARLES SCHWIETERS[etc.]Observa que un comentari pot ser d’una sola línia o multilínia.
A més, els números dels comentaris no han d’augmentar d’un en un. Per exemple, el comentari #4 és seguit immediatament pel #100.
Structure
Primary Structure Section
La secció d’estructura primària llista, entre altres coses, la seqüència d’aminoàcids de la proteïna.
El tipus principal de registre per fer això és el SEQRES.
Mirem-los a continuació:
SEQRES 1 A 33 ASP SER LYS LEU CYS TYR LEU LEU ASP GLY ILE LEU PHESEQRES 2 A 33 ILE TYR GLY VAL ILE LEU THR ALA LEU PHE LEU ARG VALSEQRES 3 A 33 LYS PHE SER ARG SER ALA ASPSEQRES 1 B 33 ASP SER LYS LEU CYS TYR LEU LEU ASP GLY ILE LEU PHESEQRES 2 B 33 ILE TYR GLY VAL ILE LEU THR ALA LEU PHE LEU ARG VALSEQRES 3 B 33 LYS PHE SER ARG SER ALA ASPEl registre SEQRES:
- Comença amb un número de sèrie que augmenta d’1 amb cada nova línia.
- Després del número de sèrie hi ha l’ID de la cadena, en aquest cas, A o B.
- Després de l’ID de la cadena hi ha el nombre de residus (aminoàcids) en aquesta cadena. Aquest número ha de ser el mateix per a cada línia de la mateixa cadena. En aquest cas, com que les cadenes A i B tenen el mateix nombre de residus (33), ambdues diuen 33.
- Cada columna posterior simplement llista els residus seqüencialment, des de l’N- fins al C-terminal (és a dir, des del terminal amino fins al carboxi).
Secondary Structure Section
La secció d’estructura secundària proporciona informació sobre l’estructura secundària de la proteïna, específicament, les hèlixs i els fulls. Podeu llegir sobre ells en detall a la pàgina d’estructura secundària de l’especificació PDB. Com podeu suposar, aquests registres donen informació sobre les α-hèlixs i els β-fulls presents en l’estructura de la proteïna.
Les estructures secundàries són determinades per diversos algoritmes utilitzats pels autors o wwPDB. Tingues en compte que aquests algoritmes no sempre coincideixen, per la qual cosa és possible que diferents bases de dades donin informació contradictòria sobre l’estructura secundària.
Connectivity Annotation Section
Aquesta secció proporciona informació sobre els enllaços o connexions presents en l’estructura de la proteïna, però que no es donen en l’estructura primària.
L’estructura 2HAC conté una d’aquestes connexions: un pont disulfur, especificat amb el registre SSBOND.
Here’s what it looks like:
SSBOND 1 CYS A 2 CYS B 2 1555 1555 2.02Aquest registre ens diu el nom de cada residu connectat (cisteïna), els seus IDs de cadena i el número de residu dins de les seves respectives cadenes.
Els següents dos camps contenen operadors de simetria, que podeu ignorar per ara. L’últim camp conté la longitud del pont disulfur en àngstroms.
Més endavant, parlarem dels registres CONECT, LINK, i CISPEP.
Crystallographic and Coordinates Section
La Secció de Transformació Cristal·logràfica i de Coordenades proporciona informació sobre l’estructura cristal·lina en què es basen les coordenades dels àtoms (amb CRYST1).
Aquesta secció també defineix el sistema de coordenades utilitzant registres ORIGXn i SCALEn.
CRYST1 Record
Moltes estructures de proteïnes es coneixen gràcies a una tècnica anomenada cristal·lografia de raigs X.
Aquesta tècnica aprofita el fet que els àtoms que formen part d’un cristall difractaran els raigs X en un patró que es pot utilitzar per elucidar les posicions relatives dels continguts del cristall dins de cada unitat repetitiva en el cristall (coneguda com a cel·la unitària).
El registre CRYST1 descriu la forma de la cel·la unitària. En estructures determinades per cristal·lografia de raigs X, aquesta descripció dona la longitud (en àngstroms) de cada aresta i els angles formats per aquestes arestes. No obstant això, l’estructura 2HAC no va ser determinada per difracció de raigs X, sinó per espectroscòpia de ressonància magnètica nuclear (espectroscòpia RMN).
En PDB, la resolució es dona en armstrongs Å, i indica quant de ben definida està cada coordenada atòmica. Com menys, millor. 2 o menys Å es considera una excel·lent resolució.
Tot i que no es van utilitzar cristalls en aquest procés, el fitxer PDB encara necessita una entrada CRYST1.
Per a 2HAC, es veu així:
CRYST1 1.000 1.000 1.000 90.00 90.00 90.00 P 1 1Aquesta és una cel·la unitària cúbica simple; cada aresta té una longitud d’1 àngstrom, tots els angles són de 90°, i el grup espacial és P 1. Qualsevol estructura no determinada per cristal·lografia utilitzarà un registre CRYST1 amb aquests valors.
ORIGXn Record
Quan una estructura de proteïna es presenta per primera vegada a l’arxiu PDB, es dona en coordenades que poden ser diferents de les utilitzades en el fitxer PDB. Les coordenades originals s’anomenen coordenades presentades, i les coordenades utilitzades en el fitxer PDB s’anomenen coordenades ortogonals. Els registres ORIGXn donen la matriu de transformació necessària per obtenir les coordenades presentades a partir de les coordenades ortogonals.
En el cas de 2HAC, ambdues coordenades són les mateixes, per la qual cosa no cal cap transformació. Per això, la matriu de transformació donada és una matriu identitat, i es veu així:
ORIGX1 1.000000 0.000000 0.000000 0.00000ORIGX2 0.000000 1.000000 0.000000 0.00000ORIGX3 0.000000 0.000000 1.000000 0.00000Observa que el nom del registre utilitza els números 1, 2 i 3 en lloc de la n en ORIGXn. n és la N-èsima fila de la matriu de transformació. Mira l’especificació ORIGXn per a més informació.
SCALEn Record
Aquest registre està relacionat amb ORIGXn. Representa la matriu de transformació necessària per passar de coordenades ortogonals a coordenades fraccionals, que són una fracció de la longitud de la cel·la unitària.
De nou, 2HAC no va ser determinada per cristal·lografia, així que es dona una matriu identitat:
SCALE1 1.000000 0.000000 0.000000 0.00000SCALE2 0.000000 1.000000 0.000000 0.00000SCALE3 0.000000 0.000000 1.000000 0.00000Coordinate Section
La secció de coordenades conté les coordenades de cada àtom per a cada model en l’estructura de la proteïna.
Aquesta secció és la més gran de qualsevol fitxer PDB. La manera més fàcil de trobar-la és buscar la primera línia que comenci amb ATOM.
Activitat Python
Crea un projecte en Python o utilitza un d’existent.
Recorda com crear-lo amb uv i editar-lo amb Pycharm.
uv init demopdbcd demopdbpycharm .Recorda com per baixar-se una proteïna de PDB des del terminal (PowerShell):
iwr https://files.rcsb.org/download/3E7Z.pdb -OutFile 3E7Z.pdbEl codi per obrir i mostrar la primera linia del fitxer que comenci per ATOM és:
with open("3E7Z.pdb") as file: for line in file: if line.startswith("ATOM"): print(line, end="") breakSi traiem el break llavors obtindrem totes les línies del fitxer que comencen per ATOM en comptes de només la primera.
Escriu un script en Python que et mostri la primera línia del fitxer 2HAC.pdb. Suposa que ja el tens descarregat.
with open("2HAC.pdb") as file: for line in file: if line.startswith("ATOM"): print(line, end="") breakEn aquesta activitat, parlarem dels registres MODEL, ATOM, i TER.
Model Record
Típicament, les proteïnes determinades per cristal·lografia només tindran un model. Com que 2HAC va ser determinada per espectroscòpia RMN, hi ha diverses conformacions de proteïnes que són consistents amb les dades de RMN. Cada conformació es registra com el seu propi model.
Un model generalment es veu així:
MODEL [model_num]ATOM 1 [...]ATOM 2 [...]ATOM 3 [...][...]ENDMDLÉs a dir, un registre de model comença amb MODEL, seguit d’una llista de registres ATOM, i acaba amb ENDMDL.
Hi ha 15 registres de model donats en 2HAC.
Escriu un script en Python que conti el número de models:
count = 0line_number = 0
with open("2HAC.pdb", 'r') as file: for line in file: line_number += 1 # comptem manualment les línies if line.startswith("MODEL"): count += 1 print(f"Line {line_number}: {line}", end="")
print("Number of models:", count)Atom Record
Un registre ATOM és d’una sola línia i apareix múltiples vegades (una per cada àtom, per cada model).
Aquí hi ha un exemple:
ATOM 2 CA ASP A -3 -24.877 1.931 -4.644 1.00 0.00 CAquest registre indica que l’àtom #2 s’anomena CA, que forma part del residu d’àcid aspàrtic a la cadena A, i el seu ID de residu és -3. Després se’ns donen les coordenades ortogonals de l’àtom (-24.877, 1.931, -4.644).
Després de les coordenades de l’àtom, tenim dos números especials: l’ocupació i el factor de temperatura (també conegut com a factor B).
L’ocupació és la proporció de temps que l’àtom passa ocupant una posició particular. Aquest camp és especialment important quan una regió molecular és molt flexible. En el cas de 2HAC, tots els àtoms (de cada model) prenen només una conformació, així que la seva ocupació és del 100% (o 1.00).
El factor B es discuteix en detall a Average and …. Els àtoms amb un factor B gran generalment corresponen a regions més flexibles de la molècula.
L’últim camp en aquest registre és el símbol elemental de l’àtom. Aquest és el símbol de l’àtom que es donaria en una taula periòdica d’elements. Com que l’àtom #2 és un àtom de carboni, el seu símbol és C.
TER Record
Com hem mencionat anteriorment, 2HAC té dues cadenes, anomenades A i B. Es dona un registre TER després que es llisten tots els àtoms d’una cadena.
Per exemple, aquest és el registre TER donat al final de la cadena A en el model 1:
ATOM 543 HB2 ASP A 30 13.952 -5.133 -14.399 1.00 0.00 HATOM 544 HB3 ASP A 30 13.434 -6.033 -12.973 1.00 0.00 HTER 545 ASP A 30ATOM 546 N ASP B -3 -24.040 -6.834 4.973 1.00 0.00 NATOM 547 CA ASP B -3 -24.918 -6.297 3.894 1.00 0.00 CEl registre TER indica el nom de l’últim residu i cadena per a l’àtom que s’acaba de donar.
Bookkeeping Section
Hi ha dos registres al final d’un fitxer PDB: MASTER i END.
El registre MASTER llista el nombre total de certs registres que apareixen:
MASTER 151 0 0 2 0 0 0 6 530 2 2 6In the above example, we can see that there are 124 REMARK records and 530 atomic coordinate records(ATOM + HETATM).
Ves a la secció MASTER de l’especificació i digues que indiquen els altres números.
La línia MASTER d’un fitxer PDB és com un resum del fitxer: cada número indica quantes línies hi ha d’un tipus concret de registre.
Això és útil per verificar ràpidament el contingut sense llegir tot el fitxer.
Segons la documentació oficial de PDB, els números corresponen habitualment a:
| Posició / número | Què indica |
|---|---|
| 1 | Nombre de línies HEADER (encapçalat) |
| 2 | Nombre de línies OBSLTE (fitxer obsolet) |
| 3 | Nombre de línies HET (heteroàtoms: ions, co-factors) |
| 4 | Nombre de línies HETNAM (noms dels heteroàtoms) |
| 5 | Nombre de línies HETSYN (sinònims dels heteroàtoms) |
| 6 | Nombre de línies FORMUL (fórmules químiques) |
| 7 | Nombre de línies HELIX (helices de la proteïna) |
| 8 | Nombre de línies SHEET (fulls beta) |
| 9 | Nombre de línies TURN (giraments) |
| 10 | Nombre de línies SITE (residus funcionals o actius) |
| 11-20 | Altres camps com XFORM, REMARK, ATOM, ANISOU, TER, HETATM, CONECT, SEQRES, CISPEP, etc. |
💡 En resum: cada número és un comptador de quantes línies d’aquell tipus hi ha al fitxer PDB.
- Per exemple, si el número corresponent a
ATOMés 483, vol dir que hi ha 483 línies ATOM. - Això et permet saber la mida i composició del fitxer sense llegir-lo completament.
És com un índex resum per la proteïna i els seus co-factors, elements secundaris i metadades.
The final record is simply END.
Escriu un script en Python que mostri la informació de la secció MASTER indicant a que correspon cada valor:
with open("2HAC.pdb", 'r') as file: for line in file: if line.startswith("MASTER"): print("Línia MASTER completa:") print(line, end="")
# Extraiem els números directament header = int(line[10:15]) obslte = int(line[15:20]) het = int(line[20:25]) hetnam = int(line[25:30]) hetsyn = int(line[30:35]) formul = int(line[35:40]) helix = int(line[40:45]) sheet = int(line[45:50]) turn = int(line[50:55]) site = int(line[55:60])
# Mostrem els valors amb f-strings print("\nResum dels comptadors:") print(f"HEADER : {header}") print(f"OBSLTE : {obslte}") print(f"HET : {het}") print(f"HETNAM : {hetnam}") print(f"HETSYN : {hetsyn}") print(f"FORMUL : {formul}") print(f"HELIX : {helix}") print(f"SHEET : {sheet}") print(f"TURN : {turn}") print(f"SITE : {site}")🐦🐦 PENDENT simplificar la solució. 🐦🐦
Més sobre el Format PDB: heterògens, lligands i aigues.
Utilitzarem eines de visualització PDB per ajudar-nos a entendre algunes característiques addicionals dels fitxers PDB.
iwr https://files.rcsb.org/download/4HG6.pdb -OutFile 4HG6.pdbL’estructura PDB 4HG6 va ser determinada per cristal·lografia de raigs X, i es veu així:
En la imatge anterior, la cadena A està acolorida en cian, la cadena B en taronja, i els àtoms d’heterògens es mostren com a esferes.
Heterògens
El format PDB es pot utilitzar per descriure residus “no estàndard”, anomenats heterògens.
La Heterogen Section i la Coordinate Section contenen informació sobre heterògens.
La secció d’heterògens de 4HG6 comença amb un registre HET a la línia 1221.
HET BGC C 1 12Aquest registre ens diu que l’heterogen #901 s’anomena BGC, forma part de la cadena A, i té 12 registres HETATM corresponents.
Unes línies més endavant, un registre HETNAM ens diu el nom complet de BGC:
HETNAM BGC BETA-D-GLUCOPYRANOSEEls registres HETATM són anàlegs als registres ATOM, excepte que donen informació sobre substàncies químiques que no són aminoàcids o nucleòtids estàndard. Per exemple, tant l’aigua com els aminoàcids modificats es consideren heterògens.
A continuació es mostra la primera entrada d’àtom heterogen:
HETATM10783 C2 BGC C 1 11.313 82.102 123.399 1.00195.30 CL’àtom té el número de sèrie 10783, s’anomena C2, forma part del residu BGC #901, i és un àtom de carboni.
Com que hi ha tants àtoms en aquesta entrada PDB, els dos primers camps (HETATM i 10783) no estan separats. Com s’ha mencionat anteriorment, això és perquè el format PDB utilitza la posició de columna per delimitar camps.
La imatge següent mostra tots els heterògens presents en 4HG6:
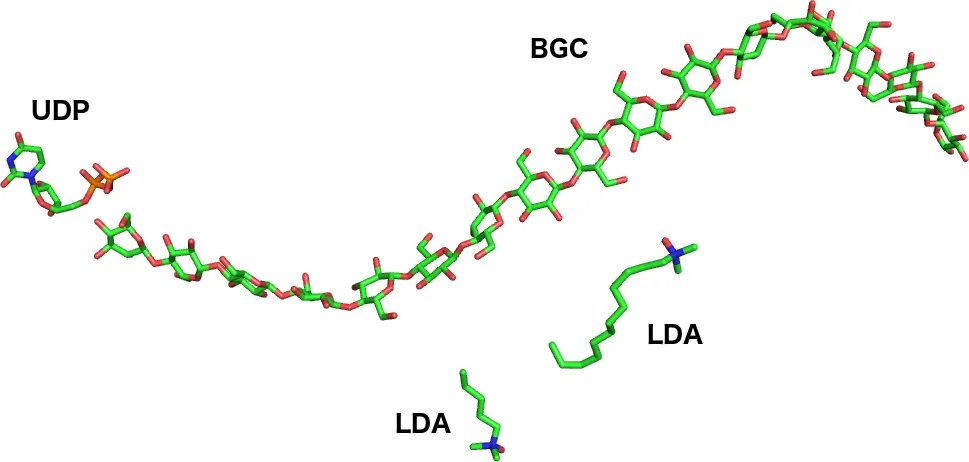
Aquí, BGC és β-D-glucosa, UDP és uridina difosfat, i LDA és òxid de lauril dimetilamina-n. Cal notar que hi ha diversos residus BGC enllaçats junts, formant un oligosacàrid d’una sola cadena. També cal notar que els àtoms d’hidrogen s’ometen d’aquesta entrada PDB. Això és perquè la resolució de raigs X ha de ser molt alta (per sota d’1Å) per veure els àtoms d’hidrogen.
Elimina les aigues de la proteina amb id 1A4W. Les linies segueixen contenen una de les dues paraules: “HOH” o “WAT”
import httpx
pdb_id = "1A4W"url = f"https://files.rcsb.org/download/{pdb_id}.pdb"
response = httpx.get(url)response.raise_for_status()
with open(f"{pdb_id}.pdb", "w") as f: f.write(response.text)
print("PDB descarregat amb aigües.")
with open(f"{pdb_id}.pdb", "r") as infile, open(f"{pdb_id}_no_water.pdb", "w") as outfile: for line in infile: # Saltamos las aguas (HOH o WAT) if line.startswith(("ATOM", "HETATM")) and line[17:20] in ("HOH", "WAT"): continue outfile.write(line)
print("Aguas eliminadas.")Connectivitat
Anteriorment hem mencionat el tipus de registre SSBOND, que dona informació sobre els enllaços disulfur.
A més d’un enllaç disulfur, 4HG6 especifica altres connexions. Els residus heterògens en 4HG6 estan connectats entre si mitjançant registres LINK:
LINK O4 BGC C 1 C1 BGC C 2 1555 1555 1.44En altres paraules, l’àtom O4 de BGC #912 està connectat a l’àtom C1 de BGC #913, i la distància d’enllaç és d’1,42 àngstroms.
El registre LINK anterior especifica una connexió entre residus (entre BGC 912 i 913). No obstant això, els heterògens també han d’especificar les connexions entre altres àtoms heterògens.
Això es fa amb el registre CONECT:
CONECT 6748 8743Aquest exemple simplement enllaça dos àtoms (#6748 i #8743).
A continuació hi ha un exemple més complicat:
CONECT10783107841078810790De nou, aquest format tan compacte es deu a l’ús de números de columna per delimitar camps, en lloc d’utilitzar un caràcter separador de camp (com una coma o espai).
Això és com es veuria el registre anterior amb espais entre cada camp:
CONECT 10783 10784 10788 10790Per entendre què està passant en aquest registre, compara’l amb la següent imatge:
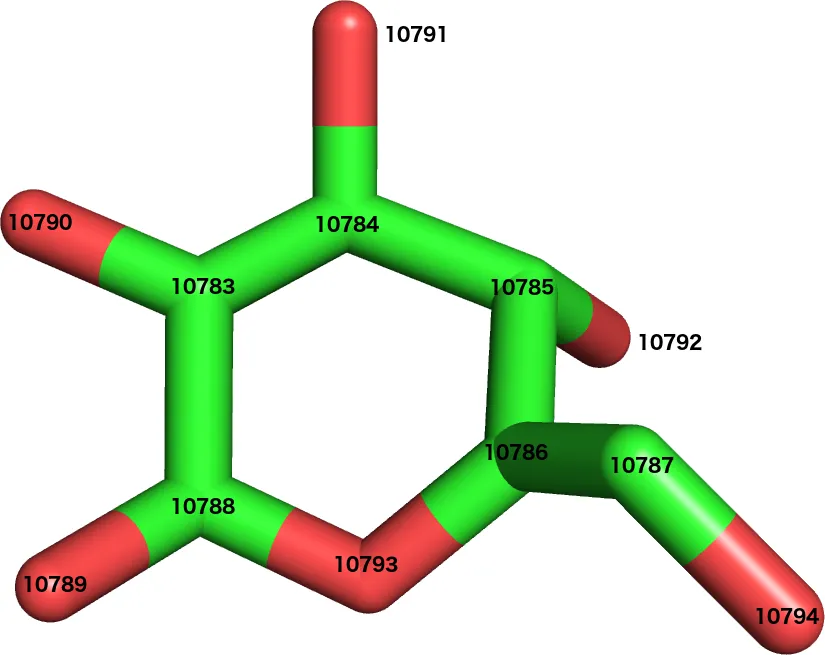
En el residu BGC anterior, cada àtom està etiquetat pel seu número de sèrie. Ara podem veure que l’àtom 10783 està connectat als àtoms 10784, 10788 i 10790.
PDB Visualization
PyMol
La forma més habitual de veure i editar les proteïnes és usar Protein - PyMOL.
Pymol is a molecular-editor program that can be used to view molecular structures like 2HAC.
iCn3D
Apart de PyMol, l’NCBI proporciona una eina molt potent per a visualitzar i treballar amb proteïnes i altres estructures en format PDB i molts altres: ICN3D.
No és tant potent ni automatizable com PyMol, però és una opció popular per a biòlegs i químics ja que no requereix saber programació i és fàcilment accesible des de qualsevol navegador.
Si obrim la pàgina ens donarà diverses opcions per visualizar en 3D les proteïnes.
O bé indicant una llista d’indentificadors (de PDB o altres) i pitjant INTRO.
Per exemple, podem posar 2 cadenes: 1HHO,4N7N;
frame “https://structure.ncbi.nlm.nih.gov/Structure/icn3d/?mmdbafid=1HH0,4N7N&bu=1”
Apart d’obtenir la animació en 3d de 2 proteïnes alhora, teniu una barra d’eines molt potent.
L’altra manera de carregar el/s fitxers PDB (o altres formats) és obrint un o més fitxers descarregats.
Provem de carregar la proteïna 6YYT del SARS-CoV-2 al portal web de l’icn3d. És la polimerasa (encarregada de replicar el virus)
Primer cal descarregar-la, amb PowerShell:
iwr https://files.rcsb.org/download/4HG6.pdb -OutFile 4HG6.pdbA Linux seria:
wget https://files.rcsb.org/download/6YYT.pdbObrim el menú principal de la pàgina, opció File i seleccionem el format.
Així és la proteïna 6YYT SARS-CoV-2:
-
Dos suports helicoïdals amb diferents tons de color blau són la cadena de plantilla d’ARN i la seva cadena de producte.
-
La major part de les cintes de color rosa és la polimerasa, que és un enzim (proteïna funcional) que fa còpies de la cadena d’ARN. Aquesta polimerasa és un objectiu atractiu per a la vacuna antiviral COVID-19.
Si voltem la molècula, podem veure les cintes grogues, verdes i taronges, que són les proteïnes víriques que ajuden a la polimerasa a mantenir-se en el camí i copiar porcions llargues de la cadena d’ARN.
Widget iCn3D
Hi ha diverses formes d’exposar la proteïna animada o una imatge estàtica (PNG) a la nostra pàgina web. S’exposa com funciona cada una a la web oficial:
Mètode senzill. Embedir el giny (widget) amb un IFrame d’HTML (tal i com faríem amb Google Maps i altres serveis)
La web proposa aquest Iframe:
<iframe allowFullScreen='true' src='https://www.ncbi.nlm.nih.gov/Structure/icn3d/?mmdbid=1tup&width=300&height=300&closepopup=1&showcommand=0&shownote=0&mobilemenu=1&showtitle=0' width='320' height='320' style='border:none'></iframe>Per a què quedi una mica millor cal decidit adaptar-lo d’aquesta manera, així es mostrarà el títol:
<main className="container mt-5"> <h2 className="text-center mt-5 fs-2">BioActivitat 6 - Estructures de proteïnes.</h2> <iframe src="https://www.ncbi.nlm.nih.gov/Structure/icn3d/?mmdbid=6YYT&showtitle=1&closepopup=1" width="100%" height="600" frameborder="0" allowfullscreen> </iframe> </main>I així ja el pots en una pàgina qualsevol.
El paràmetre de l’iframe que defineix la proteïna és mmdbid, pots provar de canviar-la per veure si et visualitza una altra proteïna; per exemple, la mmdbid=4N78.
Activitat
1.- Prova d’embedir l’ <iframe> en una de les teves pàgines web, assegura’t que es vegi bé i surti el títol de la proteïna.
<!DOCTYPE html><html lang="ca"><head> <meta charset="UTF-8"> <title>BioActivitat 6 - Estructures de proteïnes</title>
<style> body { font-family: system-ui, sans-serif; margin: 0; background: #f5f5f5; }
main { max-width: 1000px; margin: 2rem auto; padding: 1rem; background: white; border-radius: 8px; }
h1, h2, p { text-align: center; }
iframe { width: 100%; height: 600px; border: none; margin-top: 1rem; } </style></head>
<body>
<header> <h1>BioActivitat 6</h1> <p>Estructures de proteïnes amb iCn3D</p> </header>
<main> <p> Aquí podem observar una estructura proteica interactiva en 3D directament des del navegador (sense instal·lar res, pura màgia moderna ✨). Pots girar-la, fer zoom i explorar-la amb calma. </p> <p>La estructura té el codi <strong>1tup</strong></p> <iframe src="https://www.ncbi.nlm.nih.gov/Structure/icn3d/?mmdbid=6YYT&showtitle=1&closepopup=1" width="100%" height="600" frameborder="0" allowfullscreen> </iframe> </main>
<footer> <p> iCn3D és una eina proporcionada per l’<a href="https://www.ncbi.nlm.nih.gov/Structure/icn3d/icn3d.html#HowToUse">NCBI.</a> </p> </footer>
</body></html>mmCIF
El 2014, els desenvolupadors del PDB van deixar de modificar i estendre el format PDB.
Com que el format PDB utilitza la posició de columnes per delimitar camps, una de les seves principals limitacions és que no permet la descripció d’estructures que continguin més de 99.999 àtoms.
Això és perquè només es reserven 5 posicions de columna per al número de sèrie d’un àtom.
De manera similar, ja que només una columna pot descriure un identificador de cadena, cap estructura en format PDB pot tenir més de 62 cadenes (els caràcters permesos són: a-z, A-Z i 0-9).
Per aquestes i altres raons, hi ha un nou format estàndard per a estructures a l’arxiu PDB: PDBx/mmCIF, o simplement mmCIF, que significa “Macromolecular CIF”.
El nom deriva del format Crystallographic Information File (CIF), que descriu petites molècules.
En aquesta lliçó, descrivim les característiques importants del format mmCIF.
Principals estructures de proteïnes humanes
De les aproximadament 20.000 seqüències de proteïnes humanes canòniques, en data de 20 de gener de 2021, s’han determinat les estructures completes o parcials, de resolució mitjana a alta, de 7.077 proteïnes mitjançant cristal·lografia de raigs X o altres mètodes.
Quines d’aquestes proteïnes dominen el banc de dades de proteïnes (el PDB) i per què?
En aquest article, llistem les 273 principals estructures de proteïnes humanes basades en el nombre de les seves entrades PDB.
Aquest conjunt de proteïnes representa més del 40% de totes les entrades PDB humanes disponibles i representa tant les tendències passades com l’estat actual de la biologia estructural de proteïnes.