Els aminoàcids són els monòmers que componen les proteïnes.
On this page
Introducció
Els aminoàcids són els components moleculars més petits que donen forma a les proteïnes, i estan formats per àtoms de carboni, nitrogen, hidrogen i oxigen.
Així mateix, alguns, com la metionina i cisteïna, contenen sofre.
Els aminoàcids provenen del procés de transcripció de l’ADN a ARNm i la traducció posterior que efectuen els ribosomes (part fonamental de la cel·lula).
Si tens curiositat sobre aquest procés pots consultar gene expression.
Estructura
Els aminoàcids comparteixen una estructura bàsica comuna que consisteix:
- àtom central de carboni, també anomenat carboni alfa (
α) - un grup amino (–NH₂⁺)
- un grup carboxil (
COOH) - un
àtom d'hidrogen.
A més a més, tenen un grup R (un element o mol·lecula enllaçada), que és el que canvia d’un aminoàcid a un altre.
En el cas de l’aminoàcid Glicina el grup R és només un àtom d’Hidrogen (el més petit) i en canvi poden haver grups R més grans com el de la Prolina (C₅H₉NO₂).
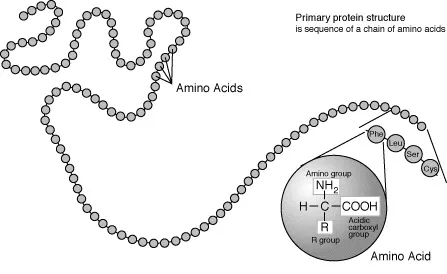
Encara que aquest dibuix els grups amino i carboxil es presenten com a neutrals per simplificar, en realitat no és l’estat en què es troben normalment els aminoàcids.
En l’ambient aquós de la cèl·lula, a pH fisiològic (7.2-7.4), tant el grup amino com el grup carboxil s’ionitzen sota condicions fisiològiques.
-
El seu grup amino recull un altre hidrogen i una càrrega positiva (
-NH3+) -
L’hidroxil en el seu grup carboxil perdria un hidrogen i guanyaria una càrrega negativa (
-COO—).
Aminoàcids proteics
Existeixen 20 aminoàcids que es troben en les proteïnes, 9 dels quals són essencials, la qual cosa es refereix al al seu cos no pot produir-los per si mateix i, per tant, han de ser obtinguts a través de la dieta.
Els 20 aminoàcids comuns (amb les seves propietats) es mostren a la taula següent.
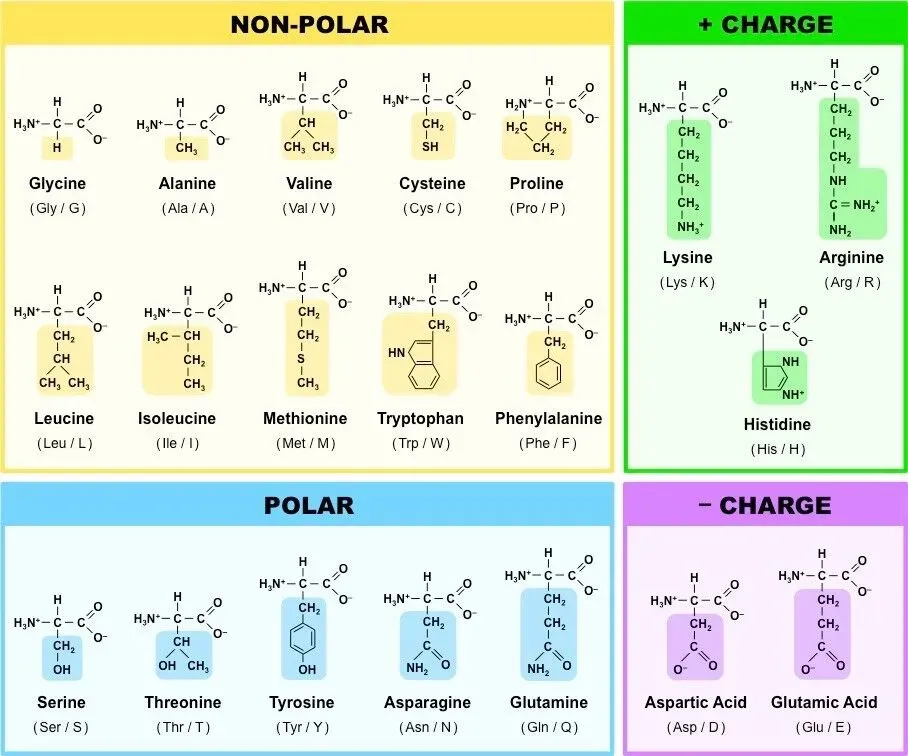
Les propietats de la cadena lateral determinen el comportament químic d’un aminoàcid (és a dir, si es considera àcid, bàsic, polar o no polar).
Per exemple, els aminoàcids com la Valina (V) i la Leucina(L) són no polars i hidrofòbics (no volen aigua), mentre que els aminoàcids com la Serina (S) i la Glutamina tenen cadenes laterals hidrofíliques (necessiten aigua) i són polars.
Alguns aminoàcids, com ara la Lisina (K) i l’Arginina (R), tenen cadenes laterals que estan carregades positivament a pH fisiològic i es consideren aminoàcids bàsics. (la Histidina (H) de vegades es considera dins aquest grup també, encara que està majoritàriament desprotonada a pH fisiològic).
L’Aspartat (D) i el Glutamat (E), per contra, estan carregats negativament a pH fisiològic i es consideren àcids.
Hidrofòbia i hidrofília
Tots els aminoàcids són o bé hidrofòbics o bé hidrofílics.
Quan una cadena d’aminoàcids entra en aigua (és a dir, a la vida real):
- els hidrofòbics fugen de l’aigua → se’n van cap a dins.
Són tots els de la categoria de No polars: Val, Leu, Ile, Met, Ala, Phe, Trp, Pro.
- els hidrofílics busquen l’aigua → es queden a fora. Són els de les altres 3 categories.
👉 Això fa que la proteïna es doblegui sola, per pura física (la natura és molt pràctica).
Grup R
Alguns altres aminoàcids tenen grups R amb propietats especials, que demostraran la seva importància quan vegem l’estructura de les proteïnes:
La prolina té un grup R que està unit al propi grup amino, formant una estructura anular. Així, la prolina és una excepció a l’estructura típica d’un aminoàcid, ja que no té el grup amino habitual NH3+.
Si creus que aquesta estructura anul·lar es veu una mica rara, tens raó: la prolina sovint provoca que les cadenes d’aminoàcids es dobleguin o torcin.
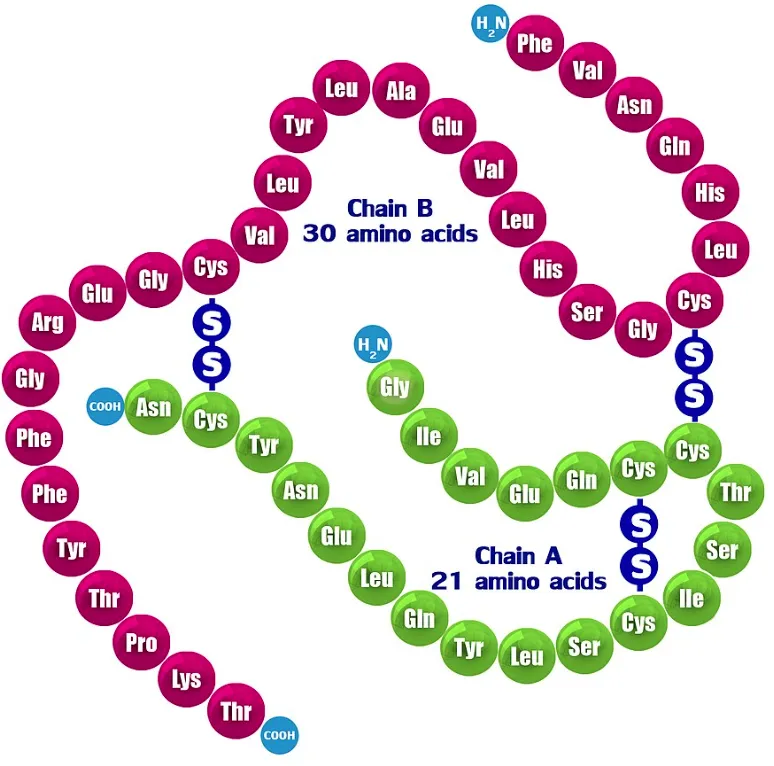
La cisteïna conté un grup tiol (-SH) i pot formar enllaços covalents amb altres cisteïnes. Aquests enllaços s’anomenen ponts de disulfur.
Anem a veure un exemple en la insulina humana:
Aminoàcids no canònics
A més dels 20 aminoàcids comuns, en certes condicions podem trobar aminoàcids “no canònics” en les proteïnes.
-
La N-formilmetionina (abreujada fMet) sol ser el primer aminoàcid de les cadenes de proteïnes produïdes per bacteris. És semblant en estructura a la metionina, però amb un grup que conté oxigen afegit al grup amino.
-
La pirrolisina s’assembla a la lisina, però té un grup addicional que conté un anell unit a l’extrem del grup R. La pirrolisina es troba principalment en les proteïnes de microorganismes anomenats arqueobacteris metanògens (productor de metà).
-
La selenocisteïna es troba en molts tipus diferents d’organismes, entre ells els éssers humans, i s’utilitza per formar les proteïnes que contenen seleni.
El seleni és altament reactiu, així que aquest aminoàcid s’ha de tractar amb cura: s’especifica al codi genètic i s’afegeix a les cadenes de proteïnes d’una manera diferent d’un aminoàcid normal.
En l’àmbit estructural, la selenocisteïna es veu igual a una cisteïna, però amb un grup –SeH en lloc del grup –SH.
Enllaç peptídic
Els aminoàcids d’un polipèptid s’uneixen als veïns mitjançant un enllaç covalent conegut com a enllaç peptídic, que es forma en una reacció de síntesi per deshidratació (condensació).
Quan els número d’AA(aminoàcids) és inferior de 10 AA es parla d’oligopèptid, quan conté entre 10 y 50 AA és un polipèptid i si el número és major normalment es tracta de proteïnes.
Tal com pots veure en el dibuix, durant la síntesi de proteïnes, el grup carboxil de l’aminoàcid al final de la cadena polipeptídica creixent reacciona amb el grup amino d’un aminoàcid entrant, alliberant una molècula d’aigua, i es crea un enllaç C-N:
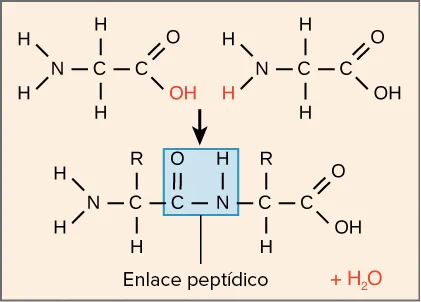
Els aminoàcids s’uneixen entre si per formar una cadena – el grup carboxil d’un aminoàcid s’uneix al grup amino d’un altre, formant un enllaç peptídic (també anomenat enllaç amina). Aquests són exemples d’enllaços químics covalents, i són estables. Quan dos aminoàcids s’uneixen, la reacció allibera una molècula d’aigua. Aquest tipus de reacció s’anomena reacció de condensació. La hidròlisi és la inversa de la reacció de condensació, i implica trencar l’enllaç peptídic utilitzant una molècula d’aigua.
En resum:
- Tenim dos aminoàcids.
- El –COO⁻ d’un reacciona amb el –NH₃⁺ de l’altre.
- Se’n va una molècula d’aigua (H₂O) → condensació.
- Es forma un enllaç peptídic (C–N).
Direccionalitat
Com has pogut veure a les imatges, la unió entre dos aminoàcids només es pot fer entre el grup amino d’un aminoàcid i el grup carboni de l’altre.
Per tant, una cadena polipeptídica té direccionalitat: té dos extrems diferents entre ells en l’àmbit químic:
-
En un extrem, el polipèptid té un grup amino lliure, anomenat amino terminal (o extrem N-terminal).
-
L’altre extrem, que té un grup carboxil lliure, es coneix com a carboxil terminal (o extrem C-terminal).
Llistat aminoàcids.
Obtenció de proteïnes.
A nivell informàtic, les cadenes d’aminoàcids que formen les proteïnes es poden trobar en diversos bancs de dades obertes, entre els quals destaquem:
PDB (Protein Data Bank)
- Base de dades internacional mantinguda principalment per RCSB PDB, amb col·laboració global.
- Emmagatzema estructures tridimensionals de proteïnes i àcids nucleics.
- Representació dels aminoàcids: 3 lletres per residu (ex.:
ALA,GLY,LYS) - Serveix per:
- Visualitzar proteïnes en 3D amb eines com PyMOL.
- Estudiar llocs actius, interaccions amb fàrmacs i química molecular.
- Analitzar formes, distàncies i orientacions dels àtoms.
- Ideal per l’estudi de fàrmacs, on la estructura és clau per entendre la funció i disseny de fàrmacs.
Treballem amb PDB a la següent sessió:
UniProt
- Base de dades internacional gestionada per un consorci europeu, nord-americà.
- Conté seqüències de proteïnes i anotacions funcionals (dominis, variants, mutacions).
- Representació dels aminoàcids: 1 lletra per aminoàcid (ex.:
MKTAYIAKQRQISFVKSHFSRQLEERLGLIEVQAN) - Serveix per:
- Identificar proteïnes i les seves característiques, per exemple la longitud o estadístiques d’aparició de cada aminoàcid.
- Comparar seqüències entre espècies o famílies.
- Treballs de programació i bioinformàtica.
- No mostra estructura 3D; només informació textual i seqüencial.
Treballem amb PDB a la següent sessió: